Le test des signes est utilisé pour comparer les médianes des observations appariés. C’est une alternative au test t pour échantillons appariés (Chapitre @ref(t-test)) et au test des rangs signés de Wilcoxon (Chapitre @ref(wilcoxon-test)) dans le cas où la distribution des différences entre les valeurs des données appariées n’est ni normale (dans le test t) ni symétrique (dans le test de Wilcoxon).
Notez que le test des signes ne fait pas d’hypothèses sur la distribution des données. Cependant, il sera probablement moins puissant que le test de Wilcoxon et le test t.
Par conséquent, si la distribution des différences entre les deux groupes appariés est symétrique, vous pourriez envisager d’utiliser le test de Wilcoxon qui est plus puissant comparé au test des signes.
Dans ce chapitre, vous apprendrez comment calculer le test des signes pour échantillons appariés en utilisant la fonction R sign_test() [paquet rstatix].
Sommaire:
Livre Apparenté
Pratique des Statistiques dans R II - Comparaison de Groupes: Variables NumériquesPrérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixoffre des fonctions R conviviales pour des analyses statistiques faciles à réaliserdatarium: contient les ensembles de données requis pour ce chapitre
Commencez par charger les packages requis suivants:
library(tidyverse)
library(rstatix)
library(ggpubr)Données de démonstration
Ici, nous utiliserons un jeu de données de démonstration mice2 [package datarium], qui contient le poids de 10 souris avant et après le traitement.
# Données larges
data("mice2", package = "datarium")
head(mice2, 3)## id before after
## 1 1 187 430
## 2 2 194 404
## 3 3 232 406# Transformez en données longues :
# rassembler les valeurs de `before` (avant) et `after` (après) dans la même colonne
mice2.long <- mice2 %>%
gather(key = "group", value = "weight", before, after)
head(mice2.long, 3)## id group weight
## 1 1 before 187
## 2 2 before 194
## 3 3 before 232Hypothèses statistiques
Le test des signes pour échantillons appariés permet d’évaluer si la médiane des différences entre les paires est statistiquement significativement différente de 0.
- Hypothèses nulles, H0 : médiane des différences appariées = 0
- Hypothèses alternatives, Ha : la médiane des différences appariées est différente de 0
Statistiques descriptives
Calcul de certaines statistiques sommaires par groupes : médiane et intervalle interquartile (IQR).
mice2.long %>%
group_by(group) %>%
get_summary_stats(weight, type = "median_iqr")## # A tibble: 2 x 5
## group variable n median iqr
## <chr> <chr> <dbl> <dbl> <dbl>
## 1 after weight 10 405 28.3
## 2 before weight 10 197. 19.2Visualisation
bxp <- ggpaired(mice2.long, x = "group", y = "weight",
order = c("before", "after"),
ylab = "Weight", xlab = "Groups")
bxp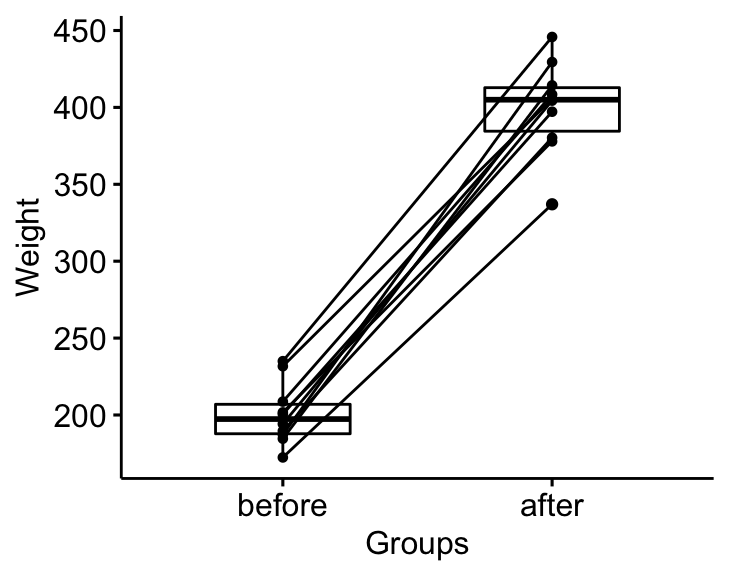
Calculs
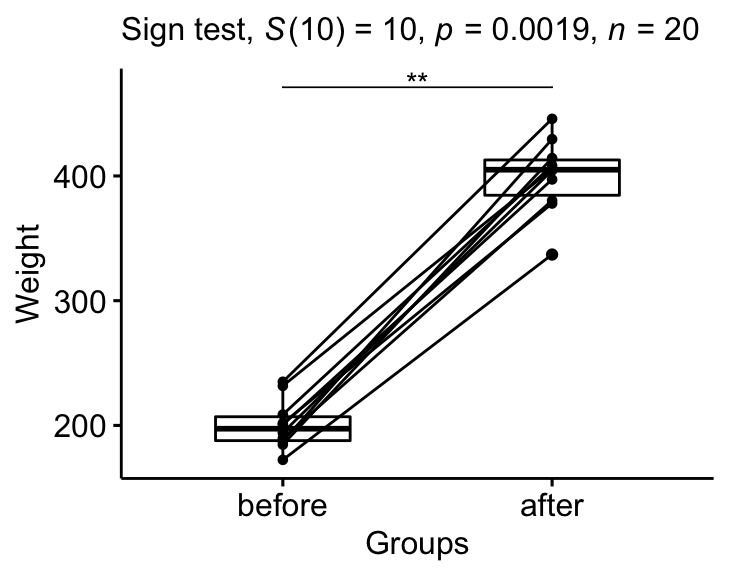
Question : Y a-t-il des changements significatifs dans le poids des souris après le traitement ?
stat.test <- mice2.long %>%
sign_test(weight ~ group) %>%
add_significance()
stat.test## # A tibble: 1 x 9
## .y. group1 group2 n1 n2 statistic df p p.signif
## <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <chr>
## 1 weight after before 10 10 10 10 0.00195 **Rapporter
Nous pourrions présenter les résultats comme suit:
Le poids médian des souris avant le traitement est significativement différent du poids médian après le traitement en utilisant le test des signes, p-value = 0,002.
stat.test <- stat.test %>% add_xy_position(x = "group")
bxp +
stat_pvalue_manual(stat.test, tip.length = 0) +
labs(
subtitle = get_test_label(stat.test, detailed= TRUE)
)
Résumé
Ce chapitre décrit comment calculer et rapporter le test des signes dans R.
Version:
 English
English



Bonjour,
Je n’arrive pas à utiliser la fonction sign_test, celle-ci fait une erreur. Sur le même jeu de données, t_test et wilcon_test fonctionnent sans problème.
Voici l’erreur :
Error in sign.test(x = c(14.5, NA, NA, 4, 13.5, 14, NA, 5, NA, 12.5, 8.5, :
‘x’ and ‘y’ must have the same length
J’ai essayé un na.omit sur les 2 colonnes pour avoir la même longueur, mais j’ai la même erreur.