Cet article décrit les hypothèses du test test et fournit des exemples de code R pour vérifier si les hypothèses sont remplies avant de calculer le test t.
Vous apprendrez les hypothèses des différents types de test t, incluant le:
- test t à échantillon unique
- test t indépendant
- test-t apparié
Sommaire:
Livre Apparenté
Pratique des Statistiques dans R II - Comparaison de Groupes: Variables NumériquesHypothèses
Le test T est un test paramétrique qui suppose certaines caractéristiques des données. Cette section montre les hypothèses faites par les différents tests t.
- Test t pour échantillon unique:
- aucune valeur aberrante significative dans les données
- les données doivent être normalement distribuées.
- T-test pour échantillon indépendant:
- aucune valeur aberrante significative dans les deux groupes
- les deux groupes d’échantillons (A et B) comparés doivent être distribués normalement.
- les variances des deux groupes ne devraient pas être significativement différentes. Cette hypothèse n’est faite que par le test t originel de Student. Elle est relaxée dans le test t du Welch.
- Test-t pour échantillons appariés:
- Aucune valeur aberrante significative dans la différence entre les deux groupes appariés
- la différence des paires doit suivre une distribution normale.
Avant d’utiliser un test paramétrique, certains tests préliminaires doivent être effectués pour s’assurer que les hypothèses du test sont respectées.
Dans les cas où les hypothèses ne sont pas respectées, des tests non paramétriques, comme le test de Wilcoxon, sont recommandés.
Vérifier les hypothèses du test t dans R
Prérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiques.datarium: contient les jeux de données requis pour ce chapitre.
Commencez par charger les packages requis suivants:
library(tidyverse)
library(ggpubr)
library(rstatix)Vérifier les hypothèses du test t à échantillon unique
Données de démonstration
Jeu de données de démonstration : mice [package datarium]. Contient le poids de 10 souris:
# Charger et inspecter les données
data(mice, package = "datarium")
head(mice, 3)## # A tibble: 3 x 2
## name weight
## <chr> <dbl>
## 1 M_1 18.9
## 2 M_2 19.5
## 3 M_3 23.1Identifier les valeurs aberrantes
Les valeurs aberrantes peuvent être facilement identifiées à l’aide des méthodes boxplot, implémentées dans la fonction R identify_outliers() [paquet rstatix].
mice %>% identify_outliers(weight)## [1] name weight is.outlier is.extreme
## <0 rows> (or 0-length row.names)Il n’y avait pas de valeurs extrêmes aberrantes.
Notez que, dans le cas où vous avez des valeurs extrêmes aberrantes, cela peut être dû à : 1) erreurs de saisie de données, erreurs de mesure ou valeurs inhabituelles.
Dans ce cas, vous pourriez envisager d’exécuter le test de Wilcoxon non paramétrique.
Vérifier l’hypothèse de normalité
L’hypothèse de normalité peut être vérifiée en calculant le test de Shapiro-Wilk. Si les données sont normalement distribuées, la p-value doit être supérieure à 0,05.
mice %>% shapiro_test(weight)## # A tibble: 1 x 3
## variable statistic p
## <chr> <dbl> <dbl>
## 1 weight 0.923 0.382Selon le résultat, la p-value est supérieure au niveau de significativité 0,05 indiquant que la distribution des données n’est pas significativement différente de la distribution normale. En d’autres termes, nous pouvons supposer que la normalité.
Vous pouvez également créer un QQ plot des données de weight. Le graphique QQ plot dessine la corrélation entre une donnée définie et la distribution normale.
ggqqplot(mice, x = "weight")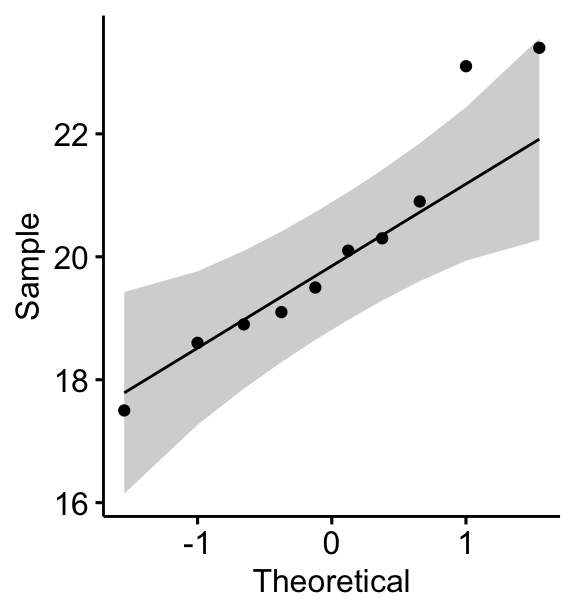
Tous les points se situent approximativement le long de la ligne de référence (45 degrés), pour chaque groupe. Nous pouvons donc supposer la normalité des données.
Notez que, si la taille de votre échantillon est supérieure à 50, le graphique de normalité QQ plot est préféré parce qu’avec des échantillons de plus grande taille, le test de Shapiro-Wilk devient très sensible même à un écart mineur par rapport à la distribution normale.
Si les données ne sont pas normalement distribuées, il est recommandé d’utiliser un test non paramétrique tel que le test de Wilcoxon à échantillon unique. Ce test est semblable au test t pour échantillon unique, mais il est axé sur la médiane plutôt que sur la moyenne.
Vérifier les hypothèses du test t indépendant
Données de démonstration
Jeu de données de démonstration : genderweight [package datarium] contenant le poids de 40 individus (20 femmes et 20 hommes).
Charger les données et afficher quelques lignes aléatoires par groupes:
# Charger les données
data("genderweight", package = "datarium")
# Afficher un échantillon des données par groupe
set.seed(123)
genderweight %>% sample_n_by(group, size = 2)## # A tibble: 4 x 3
## id group weight
## <fct> <fct> <dbl>
## 1 6 F 65.0
## 2 15 F 65.9
## 3 29 M 88.9
## 4 37 M 77.0Identifier les valeurs aberrantes par groupe
genderweight %>%
group_by(group) %>%
identify_outliers(weight)## # A tibble: 2 x 5
## group id weight is.outlier is.extreme
## <fct> <fct> <dbl> <lgl> <lgl>
## 1 F 20 68.8 TRUE FALSE
## 2 M 31 95.1 TRUE FALSEIl n’y avait pas de valeurs extrêmes aberrantes.
Vérifier la normalité par groupes
# Calculer le test d de Shapiro-Wilk par groupes
data(genderweight, package = "datarium")
genderweight %>%
group_by(group) %>%
shapiro_test(weight)## # A tibble: 2 x 4
## group variable statistic p
## <fct> <chr> <dbl> <dbl>
## 1 F weight 0.938 0.224
## 2 M weight 0.986 0.989# Dessiner un qq plot par groupe
ggqqplot(genderweight, x = "weight", facet.by = "group")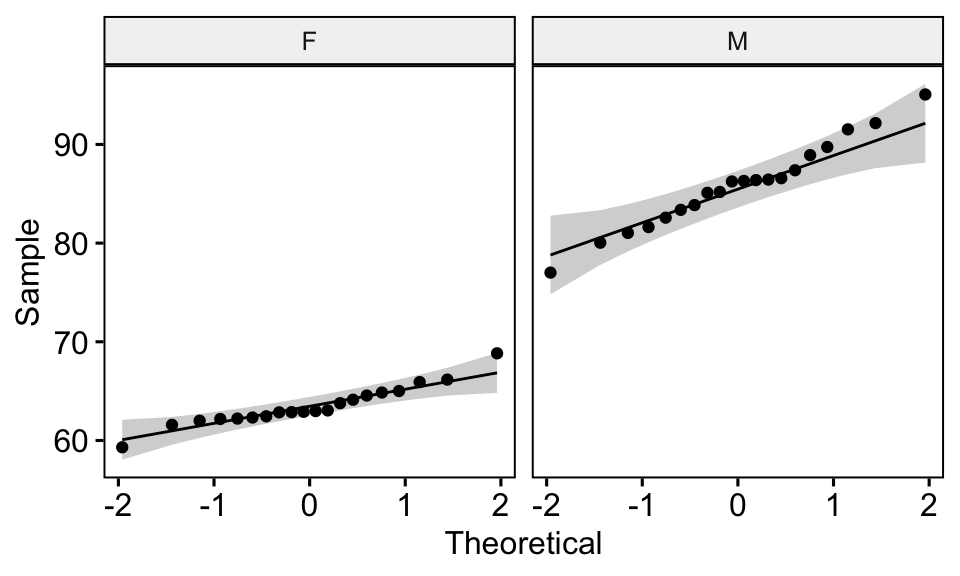
D’après les résultats ci-dessus, nous pouvons conclure que les données des deux groupes sont normalement distribuées.
Vérifier l’égalité des variances
Ceci peut être fait à l’aide du test de Levene. Si les variances des groupes sont égales, la p-value doit être supérieure à 0,05.
genderweight %>% levene_test(weight ~ group)## # A tibble: 1 x 4
## df1 df2 statistic p
## <int> <int> <dbl> <dbl>
## 1 1 38 6.12 0.0180La p-value du test de Levene est significative, ce qui suggère qu’il existe une différence significative entre les variances des deux groupes. Par conséquent, nous utiliserons le test t de Welch, qui ne suppose pas l’égalité des deux variances.
Vérifier les hypothèses du test t apparié
Données de démonstration
Ici, nous utiliserons un jeu de données de démonstration mice2 [package datarium], qui contient le poids de 10 souris avant et après le traitement.
# Format large
data("mice2", package = "datarium")
head(mice2, 3)## id before after
## 1 1 187 430
## 2 2 194 404
## 3 3 232 406# Transformez en données longues :
# rassembler les valeurs de `before` (avant) et `after` (après) dans la même colonne
mice2.long <- mice2 %>%
gather(key = "group", value = "weight", before, after)
head(mice2.long, 3)## id group weight
## 1 1 before 187
## 2 2 before 194
## 3 3 before 232Tout d’abord, commencez par calculer la différence entre les groupes:
mice2 <- mice2 %>% mutate(differences = before - after)
head(mice2, 3)## id before after differences
## 1 1 187 430 -242
## 2 2 194 404 -210
## 3 3 232 406 -174Identifier les valeurs aberrantes
mice2 %>% identify_outliers(differences)## [1] id before after differences is.outlier is.extreme
## <0 rows> (or 0-length row.names)Il n’y avait pas de valeurs extrêmes aberrantes.
Vérifier l’hypothèse de normalité
# Test de normalité de Shapiro-Wilk test pour les différences
mice2 %>% shapiro_test(differences) ## # A tibble: 1 x 3
## variable statistic p
## <chr> <dbl> <dbl>
## 1 differences 0.968 0.867# QQ plot de la différence
ggqqplot(mice2, "differences")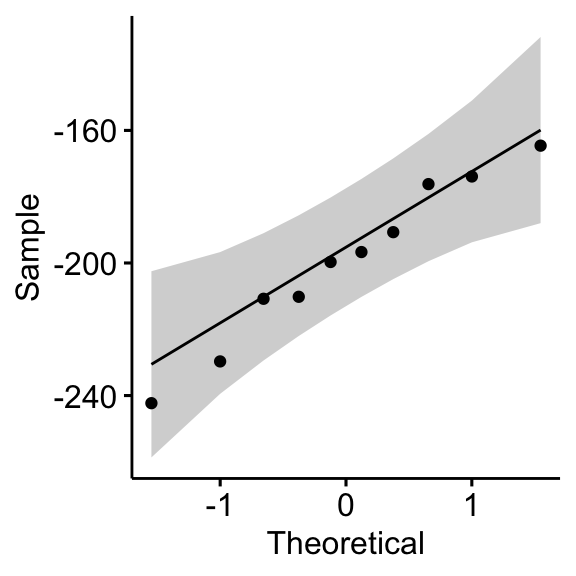
D’après les données de sortie ci-dessus, on peut supposer que les différences sont normalement distribuées.
Article apparenté
Version:
 English
English



No Comments