Différentes mesures de distance sont disponibles pour l’analyse de clustering. Cet article décrit comment effectuer un clustering dans R en utilisant la corrélation comme mesure de distance.
Contents:
Prérequis
Les packages R suivants seront utilisés:
pheatmap[package pheatmap] : Crée de jolies heatmaps.heatmap.2()[package gplots] : Une autre alternative pour créer des heatmaps.
Données de démonstration
Générer un jeu de données de démonstration:
set.seed(123)
mydata <- matrix(rnorm(200), 20, 10)
mydata[1:10, seq(1, 10, 2)] = mydata[1:10, seq(1, 10, 2)] + 3
mydata[11:20, seq(2, 10, 2)] = mydata[11:20, seq(2, 10, 2)] + 2
mydata[15:20, seq(2, 10, 2)] = mydata[15:20, seq(2, 10, 2)] + 4
colnames(mydata) = paste("Sple", 1:10, sep = "")
rownames(mydata) = paste("Gene", 1:20, sep = "")
head(mydata[, 1:4], 4)## Sple1 Sple2 Sple3 Sple4
## Gene1 2.44 -1.068 2.31 0.380
## Gene2 2.77 -0.218 2.79 -0.502
## Gene3 4.56 -1.026 1.73 -0.333
## Gene4 3.07 -0.729 5.17 -1.019Préparez vos données comme décrit à: Préparation des données et packages R pour l’analyse des clusters
Dessiner des heatmaps à l’aide de pheatmap
Par défaut, la distance euclidienne est utilisée comme mesure de dissimilarité.
library("pheatmap")
pheatmap(mydata, scale = "row")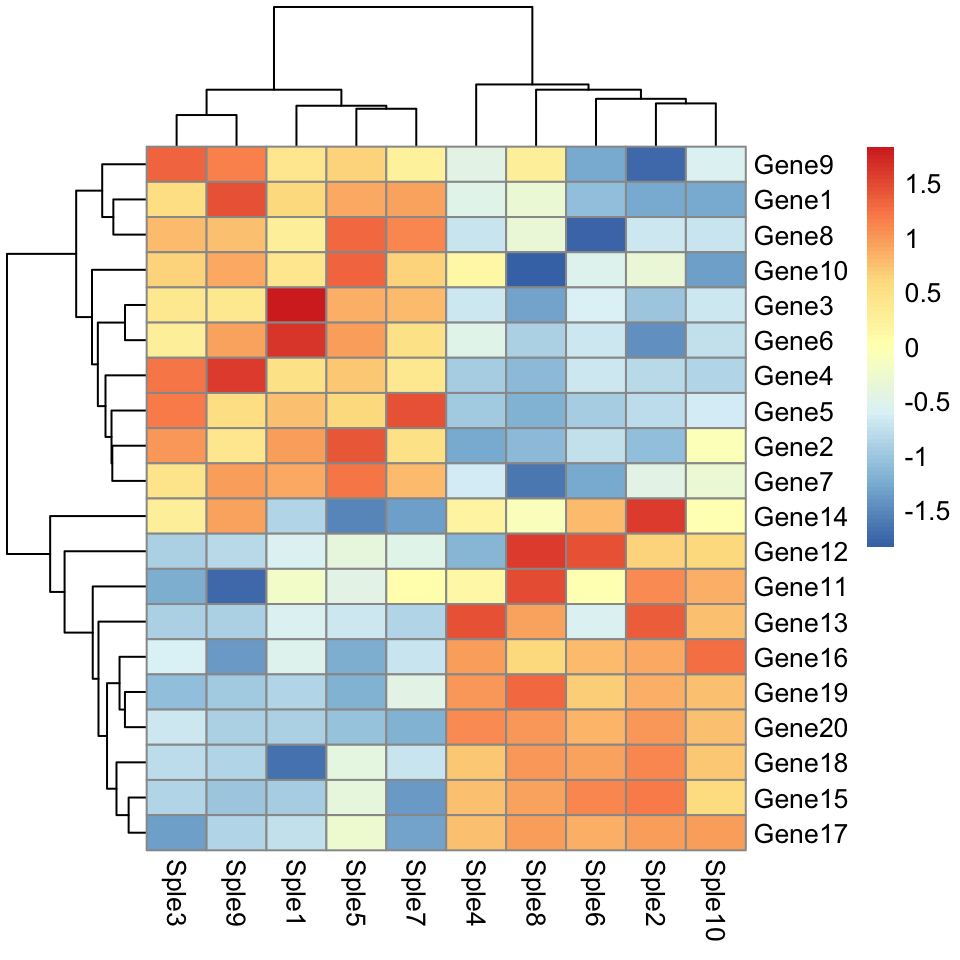
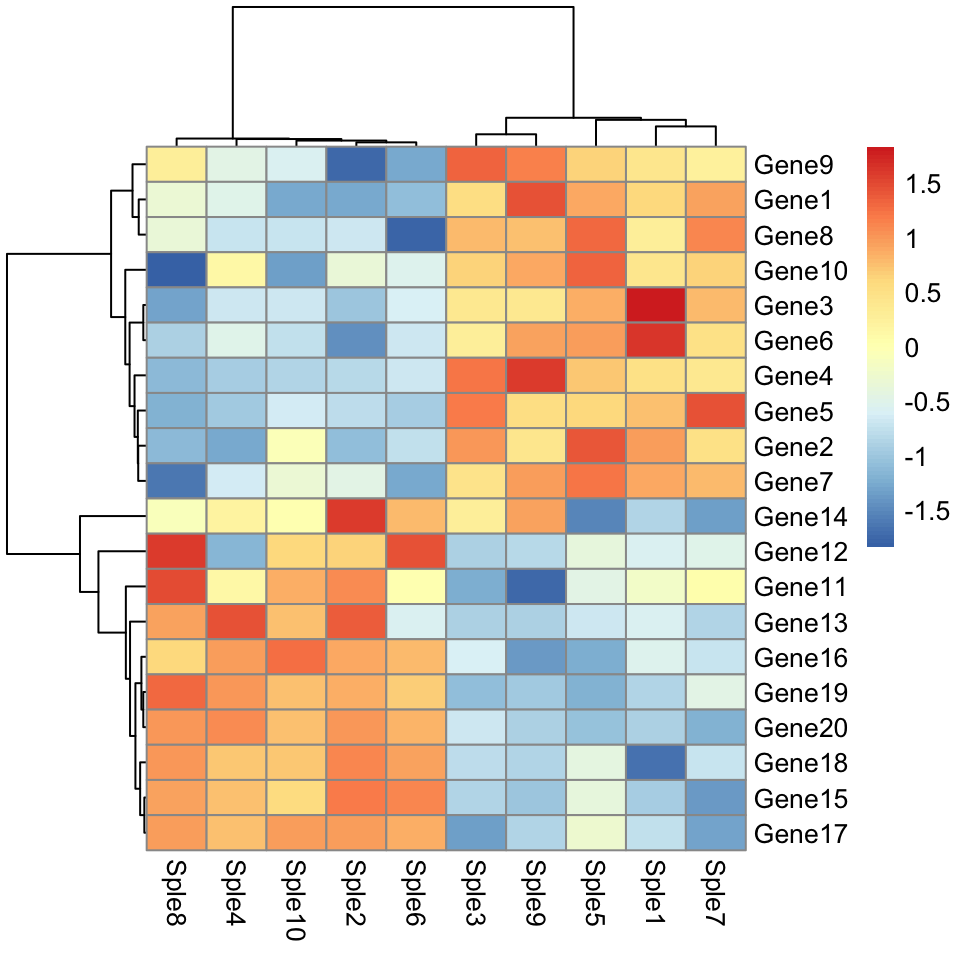
Utiliser la corrélation comme mesure de dissimilarité:
# Corrélation par paires entre les échantillons (colonnes)
cols.cor <- cor(mydata, use = "pairwise.complete.obs", method = "pearson")
# Corrélation par paires entre les lignes (gènes)
rows.cor <- cor(t(mydata), use = "pairwise.complete.obs", method = "pearson")
# Créer le heatmap
library("pheatmap")
pheatmap(
mydata, scale = "row",
clustering_distance_cols = as.dist(1 - cols.cor),
clustering_distance_rows = as.dist(1 - rows.cor)
)
Dessiner des heatmaps à l’aide de gplots
Heatmap par défaut utilisant la distance euclidienne comme mesure de dissimilarité.
library("gplots")
heatmap.2(mydata, scale = "row", col = bluered(100),
trace = "none", density.info = "none")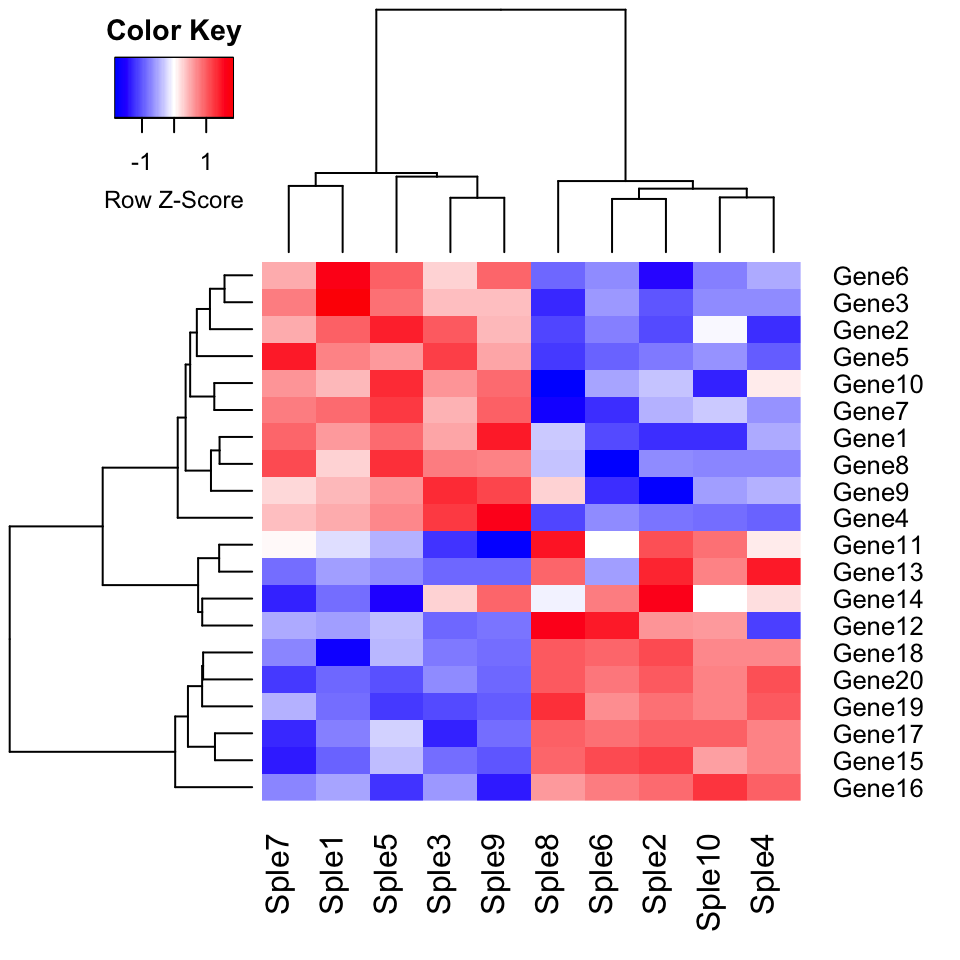
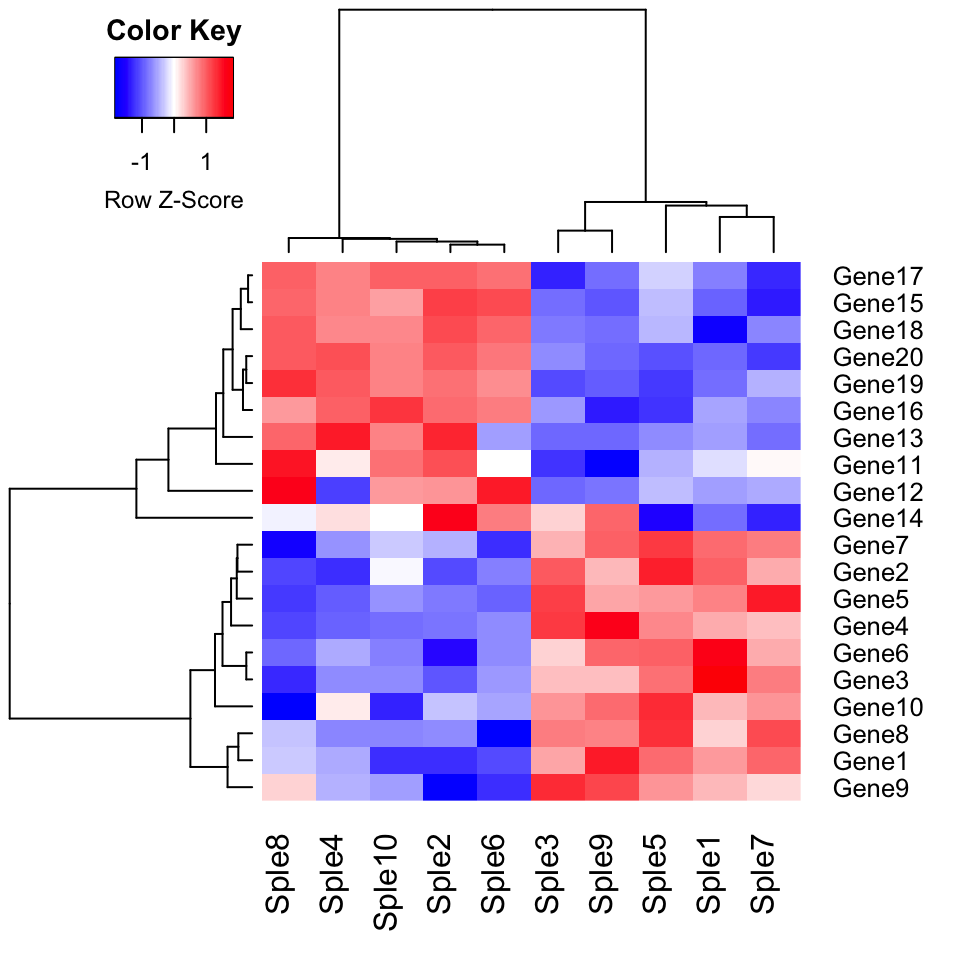
Utiliser la corrélation comme mesure de dissimilarité:
# Corrélation par paires entre les échantillons (colonnes)
cols.cor <- cor(mydata, use = "pairwise.complete.obs", method = "pearson")
# Corrélation par paires entre les lignes (gènes)
rows.cor <- cor(t(mydata), use = "pairwise.complete.obs", method = "pearson")
## Clustering par ligne et par colonne à l'aide d'une corrélation
hclust.col <- hclust(as.dist(1-cols.cor))
hclust.row <- hclust(as.dist(1-rows.cor))
# Créer le heatmap
library("gplots")
heatmap.2(mydata, scale = "row", col = bluered(100),
trace = "none", density.info = "none",
Colv = as.dendrogram(hclust.col),
Rowv = as.dendrogram(hclust.row)
)
Résumé
Dans cet article, nous présentons comment effectuer une analyse de clustering et dessiner des heatmaps dans R en utilisant les packages pheatmap et gplots
Version:
 English
English







No Comments