Cet article décrit comment ajouter des p-values sur des ggplots horizontaux en utilisant la fonction R stat_pvalue_manual() disponible dans le package R ggpubr.
- Les graphiques horizontaux peuvent être créés en utilisant la fonction
coord_flip()[dans le package ggplot2]. - Lorsque vous ajoutez des p-values à un ggplot horizontal, vous devez spécifier l’option
coord.flip = TRUEdans la fonctionstat_pvalue_manual()[dans le package ggpubr]. Les optionsvjust(ajustement vertical) ethjust(ajustement horizontal) peuvent également être spécifiées pour adapter la position des étiquettes de p-values.
Notez que, dans certains cas, les étiquettes de p-values sont partiellement cachées par la bordure supérieure du graphique. Dans ce cas, la fonction ggplot2 scale_y_continuous(expand = expansion(mult = c(0, 0.1))) peut être utilisée pour ajouter des espaces entre les étiquettes et la bordure supérieure du graphique. L’option mult = c(0, 0.1) indique que des espaces de 0% et 10% sont respectivement ajoutés en bas et en haut du graphique.
Sommaire:
Prérequis
Assurez-vous d’avoir installé les paquets R suivants:
ggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiques.
Commencez par charger les packages requis suivants:
library(ggpubr)
library(rstatix)Préparation des données
# Transformer `dose` en variable factorielle
df <- ToothGrowth
df$dose <- as.factor(df$dose)
head(df, 3)## len supp dose
## 1 4.2 VC 0.5
## 2 11.5 VC 0.5
## 3 7.3 VC 0.5Créer des graphiques simples
# Box plots
bxp <- ggboxplot(df, x = "dose", y = "len", fill = "dose",
palette = c("#00AFBB", "#E7B800", "#FC4E07"))
# Bar plots représentant la moyenne +/- l'écart-type
bp <- ggbarplot(df, x = "dose", y = "len", add = "mean_sd", fill = "dose",
palette = c("#00AFBB", "#E7B800", "#FC4E07"))Comparaisons par paires
Test statistique
Dans l’exemple suivant, nous allons effectuer un test-T en utilisant la fonction t_test() [package rstatix]. Il est également possible d’utiliser la fonction wilcox_test().
stat.test <- df %>% t_test(len ~ dose)
stat.test## # A tibble: 3 x 10
## .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 len 0.5 1 20 20 -6.48 38.0 1.27e- 7 2.54e- 7 ****
## 2 len 0.5 2 20 20 -11.8 36.9 4.40e-14 1.32e-13 ****
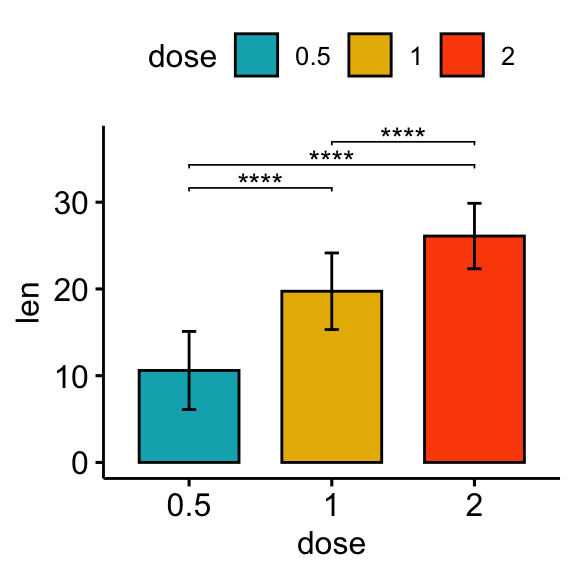
## 3 len 1 2 20 20 -4.90 37.1 1.91e- 5 1.91e- 5 ****Graphiques verticaux avec p-values
# Box plot
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp + stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01)
# Bar plot
stat.test <- stat.test %>% add_xy_position(fun = "mean_sd", x = "dose")
bp + stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01)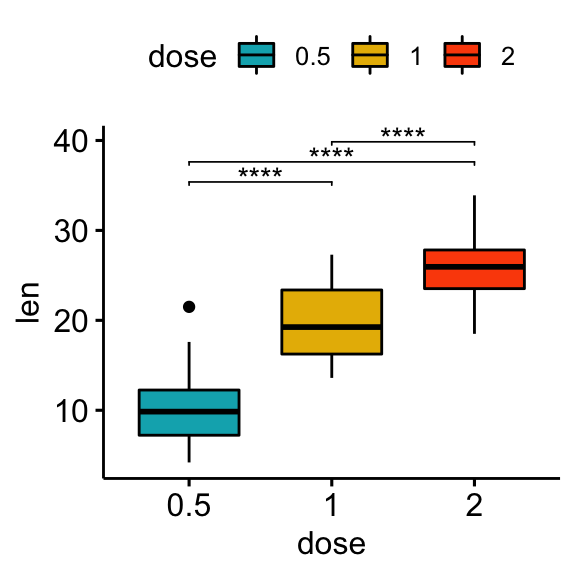
Graphiques horizontaux avec p-values
Utiliser les niveaux de significativité des p-values ajustées comme étiquettes
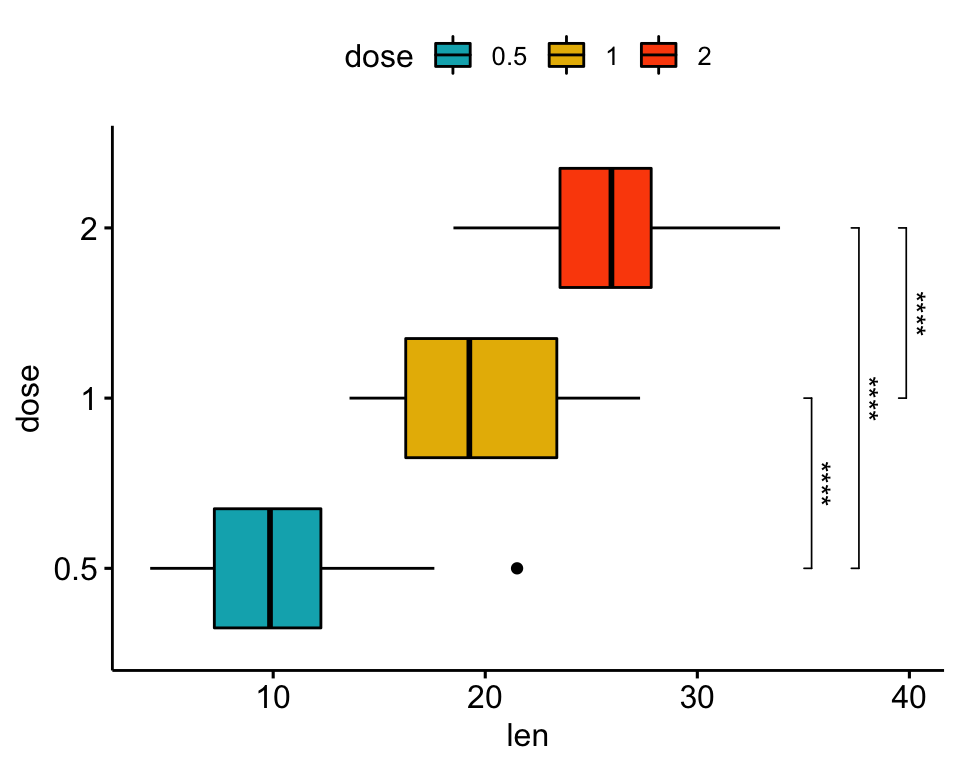
# Box plot horizontal avec p-values
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp +
stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0.01,
coord.flip = TRUE
) +
coord_flip()
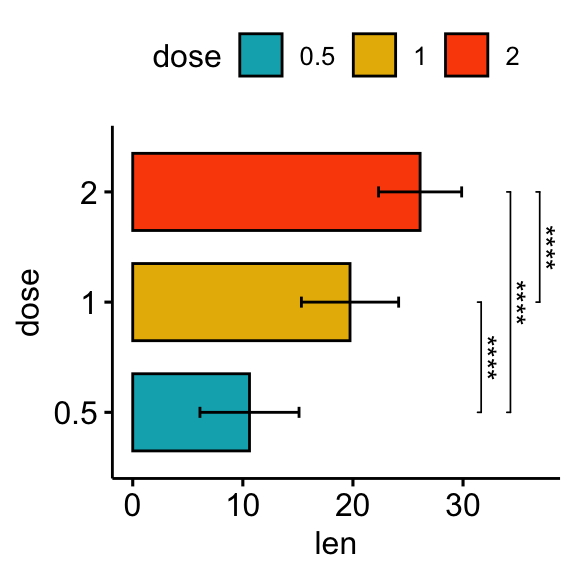
# Bar plot horizontal avec p-values
stat.test <- stat.test %>% add_xy_position(fun = "mean_sd", x = "dose")
bp + stat_pvalue_manual(
stat.test, label = "p.adj.signif", tip.length = 0.01,
coord.flip = TRUE
) +
coord_flip()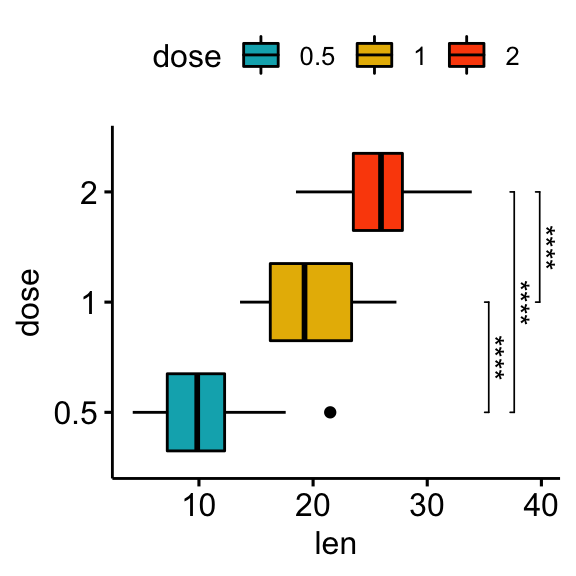
Utiliser les p-values ajustées comme étiquettes
# Utilisation de `p.adj` comme étiquettes
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp +
stat_pvalue_manual(
stat.test, label = "p.adj", tip.length = 0.01,
coord.flip = TRUE
) +
coord_flip()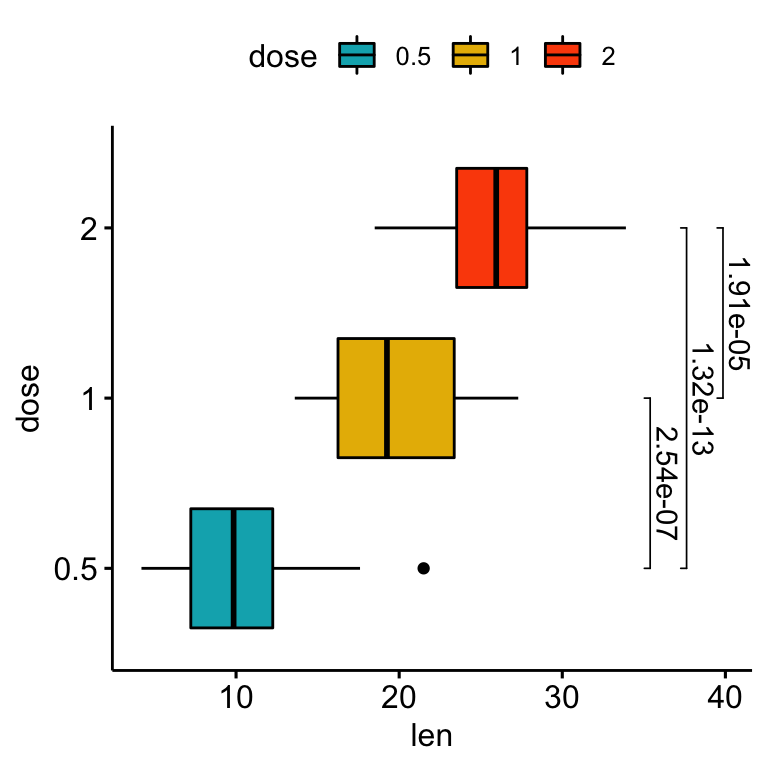
# Changer l'angle d'orientation des p-values
# Ajustez la position des p-values en utilisant vjust et hjust
# Ajoutez des espaces entre les étiquettes et la bordure du graphique
bxp +
stat_pvalue_manual(
stat.test, label = "p.adj", tip.length = 0.01,
coord.flip = TRUE, angle = 0,
hjust = 0, vjust = c(0, 1.2, 0)
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.3))) +
coord_flip() 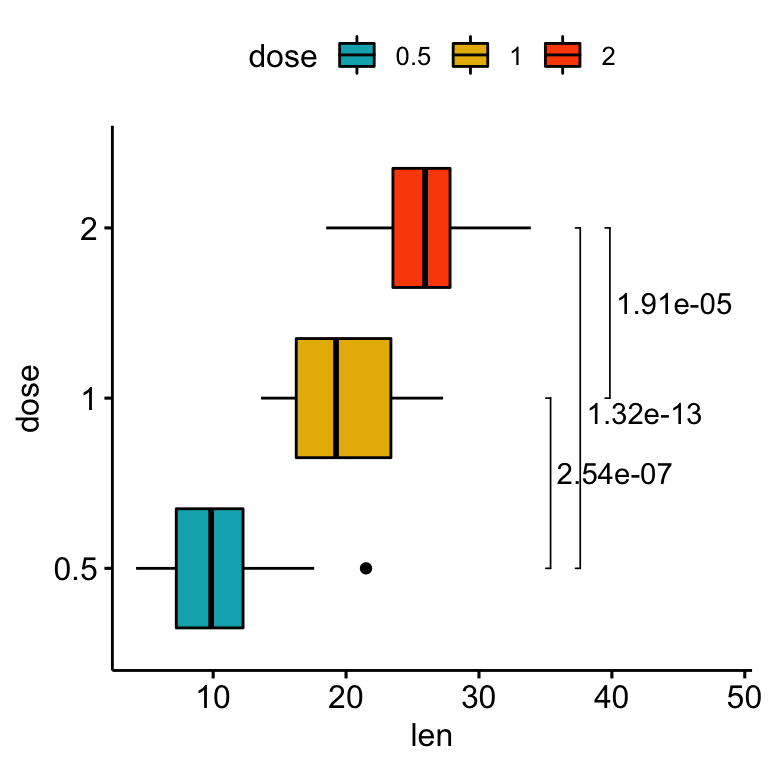
Comparaisons par rapport à un groupe de référence
Tests statistiques
stat.test <- df %>% t_test(len ~ dose, ref.group = "0.5")
stat.test## # A tibble: 2 x 10
## .y. group1 group2 n1 n2 statistic df p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 len 0.5 1 20 20 -6.48 38.0 1.27e- 7 1.27e- 7 ****
## 2 len 0.5 2 20 20 -11.8 36.9 4.40e-14 8.80e-14 ****Graphiques verticaux avec p-values
# Box plot verticale avec p-values
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp +
stat_pvalue_manual(stat.test, label = "p.adj", tip.length = 0.01) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1)))
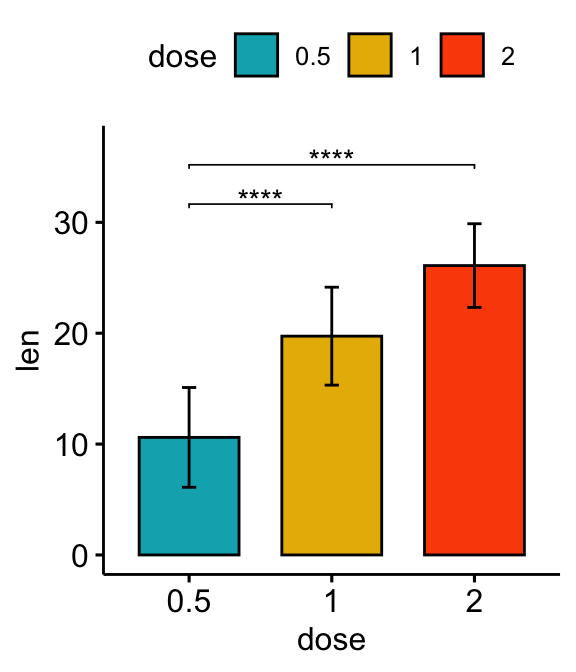
# Bar plot vertical avec p-values
stat.test <- stat.test %>% add_xy_position(fun = "mean_sd", x = "dose")
bp +
stat_pvalue_manual(stat.test, label = "p.adj.signif", tip.length = 0.01) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1)))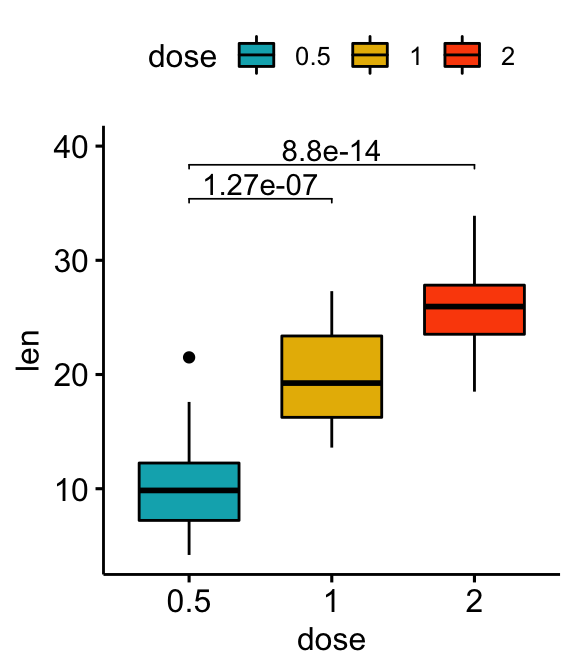
Graphiques horizontaux avec p-values
# Box plot horizontal avec p-values
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp +
stat_pvalue_manual(
stat.test, label = "p.adj", tip.length = 0.01,
coord.flip = TRUE
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1))) +
coord_flip()
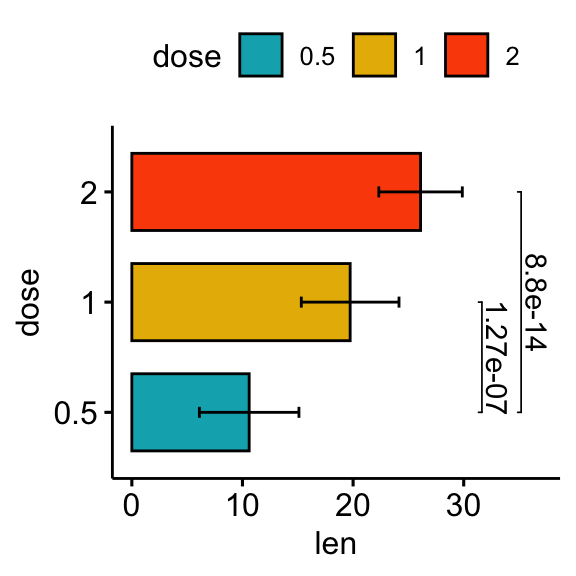
# Bar plot horizontal avec p-values
stat.test <- stat.test %>% add_xy_position(fun = "mean_sd", x = "dose")
bp +
stat_pvalue_manual(
stat.test, label = "p.adj", tip.length = 0.01,
coord.flip = TRUE
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1))) +
coord_flip()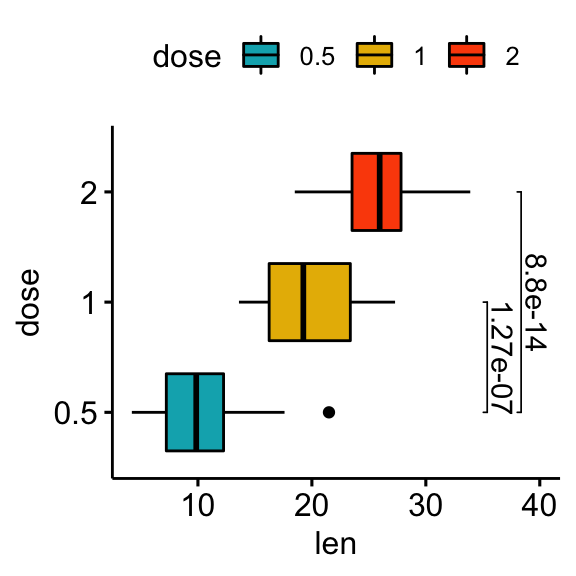
# Indiquer les p-values à la position x = "groupe2"
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp +
stat_pvalue_manual(
stat.test, label = "p = {p.adj}", tip.length = 0.01,
coord.flip = TRUE, x = "group2", hjust = 0.4
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.2))) +
coord_flip()
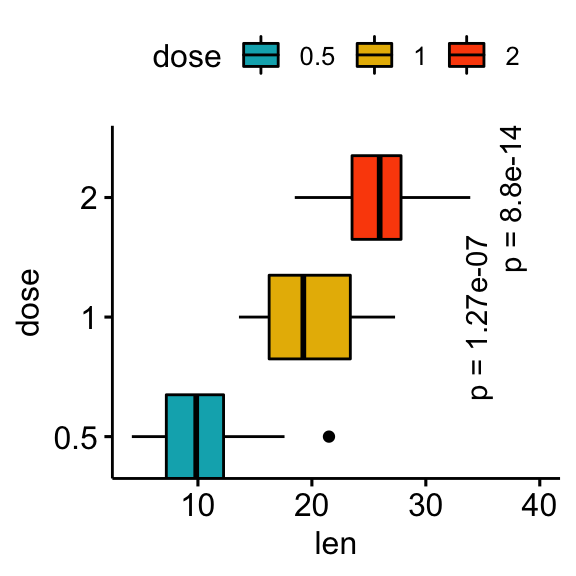
# Rendre les p-values horizontales en utilisant l'option angle
stat.test <- stat.test %>% add_xy_position(x = "dose")
bxp +
stat_pvalue_manual(
stat.test, label = "p = {p.adj}", tip.length = 0.01,
coord.flip = TRUE, x = "group2", angle = 90
) +
scale_y_continuous(expand = expansion(mult = c(0.05, 0.1))) +
scale_x_discrete(expand = expansion(mult = c(0.05, 0.3))) +
coord_flip()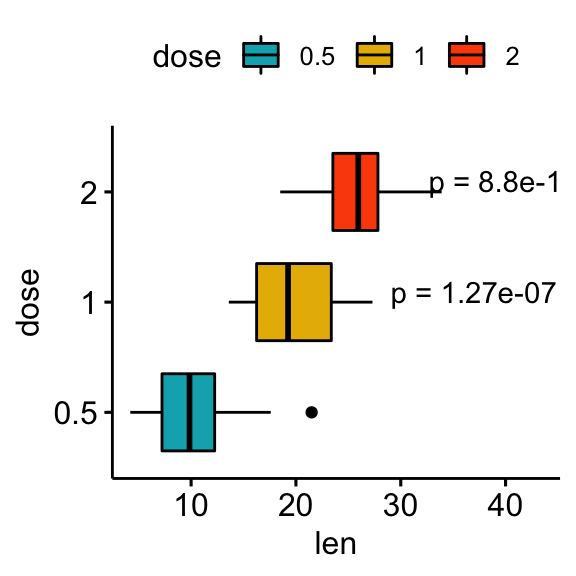
Conclusion
Cet article décrit comment ajouter des p-values sur des ggplots horizontaux en utilisant la fonction R stat_pvalue_manual() disponible dans le package R ggpubr. Voir d’autres questions apparentées fréquemment posées : ggpubr FAQ.
Version:
 English
English






No Comments