Cet article décrit comment faire un test t à échantillon unique dans R (ou dans Rstudio). Vous apprendrez à:
- Exécuter un test t à échantillon unique dans R en utilisant les fonctions suivantes :
t_test()[paquet rstatix] : le résultat est une data frame pour une visualisation graphique facile en utilisant le paquetggpubr.t.test()[paquet stats] : fonction R de base.
- Interpréter et rapporter le test t à échantillon unique
- Ajouter des p-values et des niveaux de significativité à un graphe
- Calculer et rapporter la taille de l’effet du test t à échantillon unique en utilisant le d de Cohen. La statistique “d” redéfinit la différence de moyennes comme le nombre d’écarts-types qui sépare ces moyennes. Les tailles d’effet conventionnelles des tests T, proposées par Cohen, sont : 0,2 (petit effet), 0,5 (effet modéré) et 0,8 (effet important) (Cohen 1998).
Sommaire:
Livre Apparenté
Pratique des Statistiques dans R II - Comparaison de Groupes: Variables NumériquesPrérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiques.datarium: contient les jeux de données requis pour ce chapitre.
Commencez par charger les packages requis suivants:
library(tidyverse)
library(ggpubr)
library(rstatix)Données de démonstration
Jeu de données de démonstration : mice [package datarium]. Contient le poids de 10 souris:
# Charger et inspecter les données
data(mice, package = "datarium")
head(mice, 3)## # A tibble: 3 x 2
## name weight
## <chr> <dbl>
## 1 M_1 18.9
## 2 M_2 19.5
## 3 M_3 23.1Nous voulons savoir si le poids moyen des souris diffère de 25 g (test bilatéral)
Statistiques descriptives
Calculer quelques statistiques sommaires : nombre de sujets, moyenne et sd (écart-type)
mice %>% get_summary_stats(weight, type = "mean_sd")## # A tibble: 1 x 4
## variable n mean sd
## <chr> <dbl> <dbl> <dbl>
## 1 weight 10 20.1 1.90Calculs
Utilisation de la fonction de base R
# Test t pour échantillon unique
res <- t.test(mice$weight, mu = 25)
# Affichage des résultats
res ##
## One Sample t-test
##
## data: mice$weight
## t = -8, df = 9, p-value = 2e-05
## alternative hypothesis: true mean is not equal to 25
## 95 percent confidence interval:
## 18.8 21.5
## sample estimates:
## mean of x
## 20.1Dans le résultat ci-dessus :
test la valeur de la statistique du test t (t = -8.105),dfest le degré de liberté (df= 9),p-valueest le niveau de significativité du test t (p-value = 1.99510^{-5}).conf.intest l’intervalle de confiance de la moyenne à 95% (conf.int = [18.7835, 21.4965]);sample estimatesest la valeur moyenne de l’échantillon (moyenne = 20.14).
Utilisation du paquet rstatix
Nous utiliserons le système pipe-compatible t_test() function [rstatix package], a wrapper around the R base function t.test(). Les résultats peuvent être facilement ajoutés à un graphique en utilisant le paquet Rggpubr.
stat.test <- mice %>% t_test(weight ~ 1, mu = 25)
stat.test## # A tibble: 1 x 7
## .y. group1 group2 n statistic df p
## * <chr> <chr> <chr> <int> <dbl> <dbl> <dbl>
## 1 weight 1 null model 10 -8.10 9 0.00002Les résultats ci-dessus montrent les composantes suivantes:
.y.: la variable-réponse utilisée dans le test.group1,group2: en général, les groupes comparés dans les tests par paires. Ici, nous avons le modèle nul (test pour échantillon unique).statistic: statistique du test (valeur t) utilisée pour calculer la p-value.df: degrés de liberté.p: p-value.
Vous pouvez obtenir un résultat détaillé en spécifiant l’option detailed = TRUE dans la fonction t_test().
mice %>% t_test(weight ~ 1, mu = 25, detailed = TRUE)## # A tibble: 1 x 12
## estimate .y. group1 group2 n statistic p df conf.low conf.high method alternative
## * <dbl> <chr> <chr> <chr> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <chr> <chr>
## 1 20.1 weight 1 null model 10 -8.10 0.00002 9 18.8 21.5 T-test two.sidedInterprétation
La p-value du test est 210^{-5}, ce qui est inférieure au seuil de significativité alpha = 0,05. Nous pouvons conclure que le poids moyen des souris est significativement différent de 25g avec une p-value = 210^{-5}.
Taille de l’effet
Pour calculer la taille de l’effet, appelée d de Cohen, du test t pour échantillon unique, vous devez diviser la différence moyenne par l’écart type de la différence, comme indiqué ci-dessous. Notez que, ici: sd(x-mu) = sd(x).
La formule du d de Cohen:
\[
d = \frac{m-\mu}{s}
\]
- \(m\) est la moyenne de l’échantillon
- \(s\) est l’écart-type de l’échantillon avec les degrés de liberté \(n-1\)
- \(\mu\) est la moyenne théorique à laquelle la moyenne de notre échantillon est comparée (la valeur par défaut est mu = 0).
Calculs:
mice %>% cohens_d(weight ~ 1, mu = 25)## # A tibble: 1 x 6
## .y. group1 group2 effsize n magnitude
## * <chr> <chr> <chr> <dbl> <int> <ord>
## 1 weight 1 null model -2.56 10 largeRappelons que les taille de l’effet conventionnelles du test t, proposé par Cohen J. (1998), sont : 0,2 (petit effet), 0,5 (effet modéré) et 0,8 (effet important) (Cohen 1998). Comme la taille de l’effet, d, est de 2,56, vous pouvez conclure qu’il y a un effet important.
Rapports
Nous pourrions rapporter le résultat comme suit:
Un test t pour échantillon unique a été calculé pour déterminer si le poids moyen des souris incluses était différent du poids moyen normal de la population (25 g).
Le poids moyen mesuré des souris (20,14 +/- 1,94) était statistiquement significativement inférieur au poids moyen normal de la population 25 (t(9) = -8,1, p < 0,0001, d = 2,56) ; où t(9) est une notation courte pour une statistique t qui a 9 degrés de liberté.
Les résultats peuvent être visualisés à l’aide d’un box plot ou d’un diagramme de densité.
Box Plot
Créer un boxplot pour visualiser la distribution du poids des souris. Ajoutez également des points jitter pour montrer les observations individuelles. Le gros point représente le point moyen.
# Créer le box-plot
bxp <- ggboxplot(
mice$weight, width = 0.5, add = c("mean", "jitter"),
ylab = "Weight (g)", xlab = FALSE
)
# Ajouter les niveaux de significativité
bxp + labs(subtitle = get_test_label(stat.test, detailed = TRUE))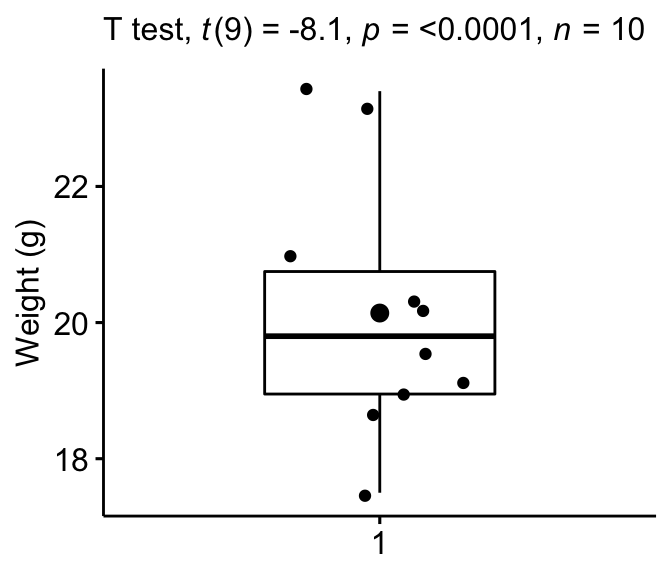
Diagramme de densité
Créer un graphe de densité avec p-value:
- La ligne rouge correspond à la moyenne observée
- La ligne bleue correspond à la moyenne théorique
ggdensity(mice, x = "weight", rug = TRUE, fill = "lightgray") +
scale_x_continuous(limits = c(15, 27)) +
stat_central_tendency(type = "mean", color = "red", linetype = "dashed") +
geom_vline(xintercept = 25, color = "blue", linetype = "dashed") +
labs(subtitle = get_test_label(stat.test, detailed = TRUE))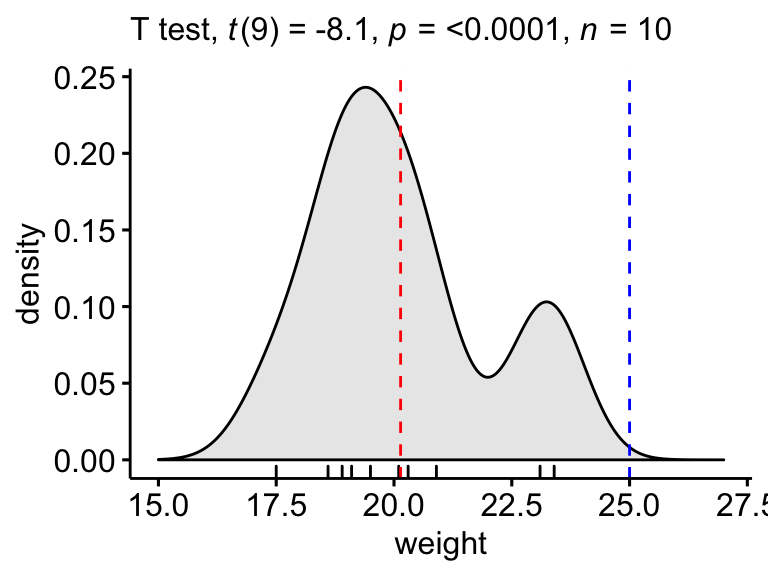
Résumé
Cet article montre comment effectuer le t-test à échantillon unique dans R/Rstudio en utilisant deux manières différentes : la fonction de base R t.test() et la fonction t_test() dans le paquet rstatix. Nous décrivons également comment interpréter et communiquer les résultats du test t.
References
Cohen, J. 1998. Statistical Power Analysis for the Behavioral Sciences. 2nd ed. Hillsdale, NJ: Lawrence Erlbaum Associates.
Version:
 English
English



No Comments