Le test de Kruskal-Wallis est une alternative non paramétrique au test ANOVA à un facteur. Il étend le test de Wilcoxon à deux échantillons dans les cas où il y a plus de deux groupes à comparer. Il est recommandé lorsque les hypothèses du test ANOVA, à un facteur, ne sont pas respectées.
Ce chapitre décrit comment calculer le test Kruskal-Wallis à l’aide du logiciel R. Vous apprendrez également comment calculer la taille de l’effet à partir de la statistique H de kruskal-Wallis.
Sommaire:
Livre Apparenté
Pratique des Statistiques dans R II - Comparaison de Groupes: Variables NumériquesPrérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixoffre des fonctions R conviviales pour des analyses statistiques faciles à réaliser.
Charger les packages:
library(tidyverse)
library(ggpubr)
library(rstatix)Préparation des données
Ici, nous utiliserons le jeu de données intégré dans R appelé PlantGrowth. Il contient le poids des plantes obtenues sous un contrôle et deux conditions de traitement différentes.
set.seed(1234)
PlantGrowth %>% sample_n_by(group, size = 1)## # A tibble: 3 x 2
## weight group
## <dbl> <fct>
## 1 5.58 ctrl
## 2 6.03 trt1
## 3 4.92 trt2- Réorganiser les niveaux de groupes:
PlantGrowth <- PlantGrowth %>%
reorder_levels(group, order = c("ctrl", "trt1", "trt2"))statistiques descriptives
Calculer des statistiques descriptives par groupe:
PlantGrowth %>%
group_by(group) %>%
get_summary_stats(weight, type = "common")## # A tibble: 3 x 11
## group variable n min max median iqr mean sd se ci
## <fct> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 ctrl weight 10 4.17 6.11 5.16 0.743 5.03 0.583 0.184 0.417
## 2 trt1 weight 10 3.59 6.03 4.55 0.662 4.66 0.794 0.251 0.568
## 3 trt2 weight 10 4.92 6.31 5.44 0.467 5.53 0.443 0.14 0.317Visualisation
Créez un boxplot de weight en group:
ggboxplot(PlantGrowth, x = "group", y = "weight")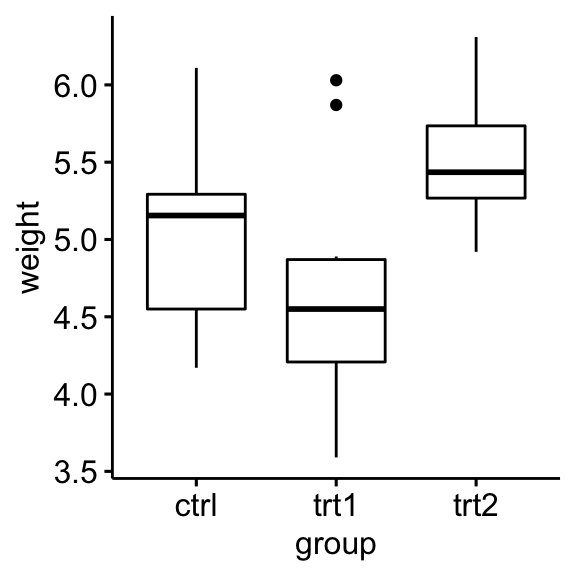
Calculs
Question : Nous voulons savoir s’il y a une différence significative entre les poids moyens des plantes dans les 3 conditions expérimentales.
Nous utiliserons la fonction kruskal_test() [package rstatix], un wrapper autour de la fonction R de base kruskal.test().
res.kruskal <- PlantGrowth %>% kruskal_test(weight ~ group)
res.kruskal## # A tibble: 1 x 6
## .y. n statistic df p method
## * <chr> <int> <dbl> <int> <dbl> <chr>
## 1 weight 30 7.99 2 0.0184 Kruskal-WallisTaille de l’effet
L’eta-carré, basé sur la statistique H, peut être utilisé comme mesure de la taille de l’effet du test Kruskal-Wallis. Il se calcule comme suit : eta2[H] = (H - k + 1)/(n - k) ; où H est la valeur obtenue dans le test de Kruskal-Wallis ; k est le nombre de groupes ; n est le nombre total d’observations (M. T. Tomczak and Tomczak 2014).
L’estimation eta-carré est comprise entre 0 et 1; la valeur multipliée par 100 indique le pourcentage de variance de la variable dépendante expliqué par la variable indépendante.
Les valeurs d’interprétation couramment utilisées dans la littérature sont : 0,01- < 0,06 (petit effet), 0,06 - < 0,14 (effet modéré) et >= 0,14 (effet important).
PlantGrowth %>% kruskal_effsize(weight ~ group)## # A tibble: 1 x 5
## .y. n effsize method magnitude
## * <chr> <int> <dbl> <chr> <ord>
## 1 weight 30 0.222 eta2[H] largeUne grande taille d’effet est détectée, eta2[H] = 0.22.
Comparaisons multiples par paires
D’après les résultats du test de Kruskal-Wallis, nous savons qu’il existe une différence significative entre les groupes, mais nous ne savons pas quelles paires de groupes sont différentes.
Un test de Kruskal-Wallis significatif est généralement suivi d’un test de Dunn pour identifier les groupes différents. Il est également possible d’utiliser le test de Wilcoxon pour calculer des comparaisons par paires entre les niveaux de groupe avec des corrections pour les tests multiples.
Par rapport au test de Wilcoxon, le test de Dunn tient compte du classement utilisé par le test de Kruskal-Wallis. Il fait aussi des ajustements pour les exaequos.
- Comparaisons par paires à l’aide du test de Dunn:
# Comparaisons par paires
pwc <- PlantGrowth %>%
dunn_test(weight ~ group, p.adjust.method = "bonferroni")
pwc## # A tibble: 3 x 9
## .y. group1 group2 n1 n2 statistic p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <chr>
## 1 weight ctrl trt1 10 10 -1.12 0.264 0.791 ns
## 2 weight ctrl trt2 10 10 1.69 0.0912 0.273 ns
## 3 weight trt1 trt2 10 10 2.81 0.00500 0.0150 *- Comparaisons par paires à l’aide du test de Wilcoxon:
pwc2 <- PlantGrowth %>%
wilcox_test(weight ~ group, p.adjust.method = "bonferroni")
pwc2## # A tibble: 3 x 9
## .y. group1 group2 n1 n2 statistic p p.adj p.adj.signif
## * <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <chr>
## 1 weight ctrl trt1 10 10 67.5 0.199 0.597 ns
## 2 weight ctrl trt2 10 10 25 0.063 0.189 ns
## 3 weight trt1 trt2 10 10 16 0.009 0.027 *La comparaison par paires montre que seuls trt1 et trt2 sont significativement différents (test de Wilcoxon, p = 0,027).
Rapporter
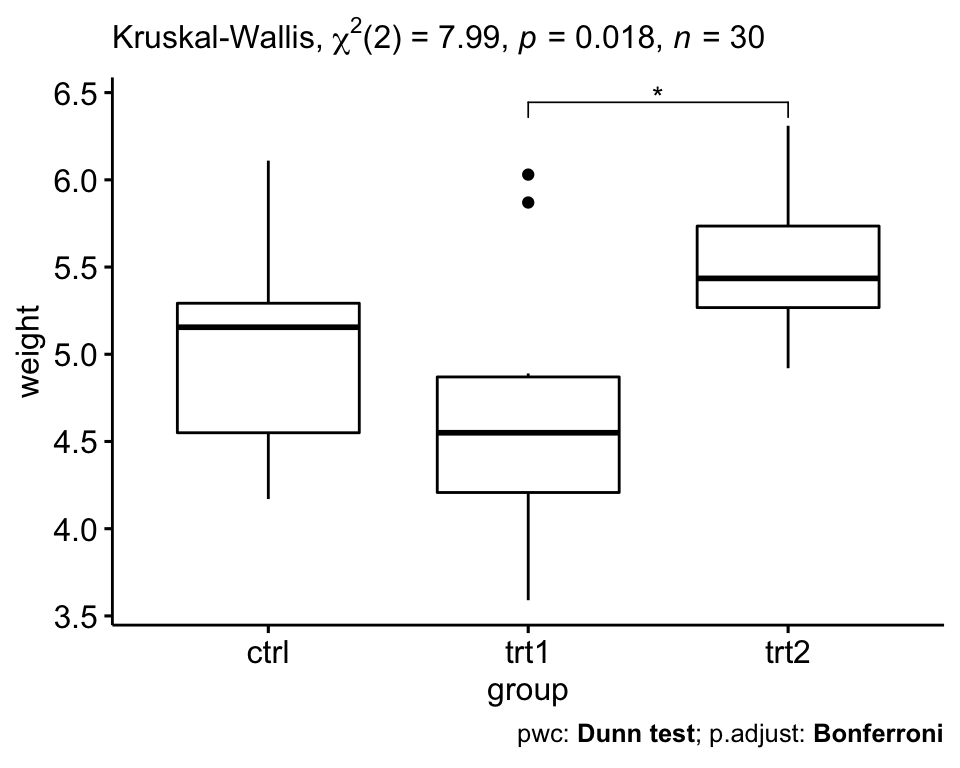
Il y avait des différences statistiquement significatives entre les groupes de traitement, évaluées à l’aide du test de Kruskal-Wallis (p = 0,018). Le test de Wilcoxon par paires entre les groupes a montré que seule la différence entre les groupes trt1 et trt2 était significative (test de Wilcoxon, p = 0,027)
# Visualisation : Boxplots avec p-values
pwc <- pwc %>% add_xy_position(x = "group")
ggboxplot(PlantGrowth, x = "group", y = "weight") +
stat_pvalue_manual(pwc, hide.ns = TRUE) +
labs(
subtitle = get_test_label(res.kruskal, detailed = TRUE),
caption = get_pwc_label(pwc)
)
References
Tomczak, Maciej T., and Ewa Tomczak. 2014. “The Need to Report Effect Size Estimates Revisited. an Overview of Some Recommended Measures of Effect Size.” Trends in SportSciences.
Version:
 English
English



No Comments