Le test de Wilcoxon est une alternative non paramétrique au test t pour comparer deux moyennes. C’est particulièrement recommandé dans une situation où les données ne sont pas normalement distribuées.
Comme le test t, le test de Wilcoxon se présente sous deux formes : tests sur échantillon unique et sur deux échantillons. Ils sont utilisés dans plus ou moins les mêmes situations que les tests t correspondants.
Notez que la taille de l’échantillon doit être d’au moins 6. Autrement, le test de Wilcoxon ne peut devenir significatif.
Dans ce chapitre, vous apprendrez comment calculer les différents types de tests de Wilcoxon dans R, notamment:
- Test des rangs signés de Wilcoxon sur échantillon unique
- Test de la somme des rangs de Wilcoxon et
- Test des rangs signés de Wilcoxon sur échantillons appariés
- Vérifier les hypothèses du test de Wilcoxon
- Calculer et rapporter la taille de l’effet du test de Wilcoxon (valeur r).
La taille de l’effet r est calculée comme étant la statistique Z divisée par la racine carrée de la taille de l’échantillon (N) (Z/sqrt(N)). La valeur Z est extraite soit de coin::wilcoxsign_test() (cas d’un test à un ou deux échantillons) ou coin::wilcox_test() (cas d’un test indépendant à deux échantillons).
Notez que N correspond à la taille totale de l’échantillon pour le test d’échantillons indépendants et au nombre total de paires pour le test d’échantillons appariés. La valeur r varie de 0 à près de 1. Les valeurs d’interprétation de “r” couramment utilisées dans la littérature publiée sont : 0,10 - < 0,3 (petit effet), 0,30 - < 0,5 (effet modéré) et >= 0,5 (effet important).
Nous utiliserons la fonction wilcox_test() [paquet rstatix], qui est compatible avec les pipes.
Sommaire:
Livre Apparenté
Pratique des Statistiques dans R II - Comparaison de Groupes: Variables NumériquesPrérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiquesdatarium: contient les ensembles de données requis pour ce chapitre
Commencez par charger les packages requis suivants:
library(tidyverse)
library(rstatix)
library(ggpubr)Test des rangs signés de Wilcoxon sur échantillon unique
Le test des rangs signés de Wilcoxon sur échantillon unique est utilisé pour évaluer si la médiane de l’échantillon est égale à une valeur standard ou théorique connue. Il s’agit d’un équivalent non paramétrique du test t sur échantillon unique.
Données de démonstration
Jeu de données de démonstration : mice [package datarium]. Contient le poids de 10 souris:
# Charger et inspecter les données
data(mice, package = "datarium")
head(mice, 3)## # A tibble: 3 x 2
## name weight
## <chr> <dbl>
## 1 M_1 18.9
## 2 M_2 19.5
## 3 M_3 23.1Statistiques descriptives
Calculer la médiane et l’écart interquartile (IQR):
mice %>% get_summary_stats(weight, type = "median_iqr")## # A tibble: 1 x 4
## variable n median iqr
## <chr> <dbl> <dbl> <dbl>
## 1 weight 10 19.8 1.8Visualisation
Créez un boxplot pour visualiser la distribution du poids des souris. Ajoutez également des points jitter pour montrer les observations individuelles. Le gros point représente le point moyen.
bxp <- ggboxplot(
mice$weight, width = 0.5, add = c("mean", "jitter"),
ylab = "Weight (g)", xlab = FALSE
)
bxp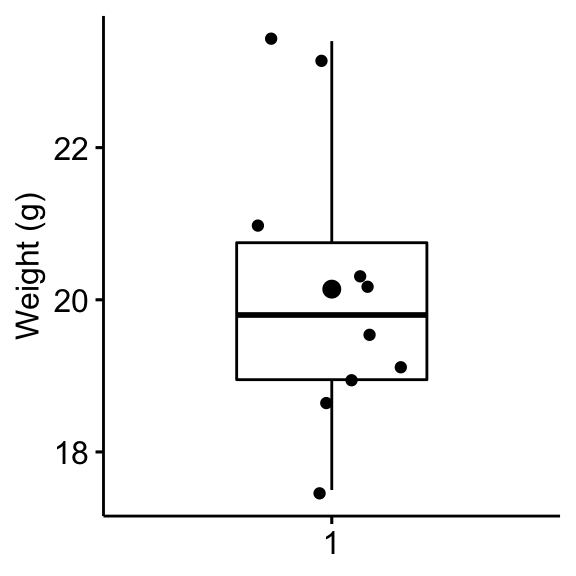
Hypothèses et tests préliminaires
Le test de Wilcoxon suppose que les données sont distribuées symétriquement autour de la médiane. Ceci peut être vérifié par une inspection visuelle à l’aide d’un histogramme et d’une distribution de densité.
Créer un histogramme : Comme nous n’avons que 10 individus dans nos données, nous spécifions l’option bins = 4 au lieu de 30 (par défaut).
gghistogram(mice, x = "weight", y = "..density..",
fill = "steelblue",bins = 4, add_density = TRUE)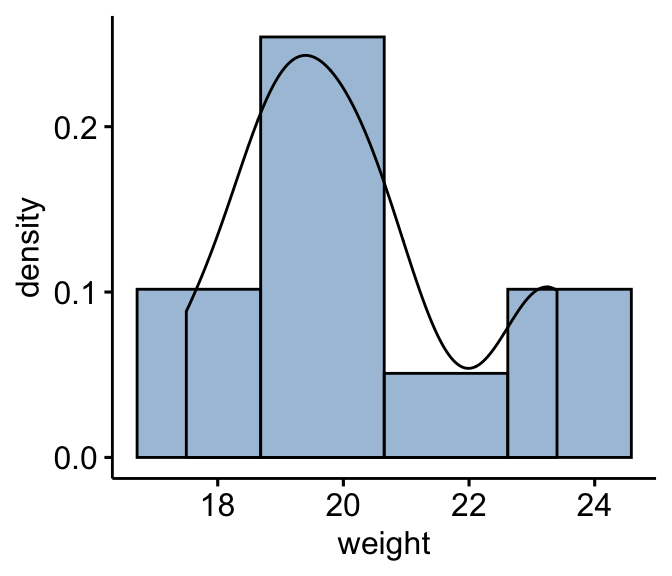
D’après le graphique ci-dessus, on peut voir que les données de poids (weight) sont approximativement symétriques (il ne faut pas s’attendre à ce qu’elles soient parfaites, en particulier lorsque le nombre d’échantillons dans votre étude est inférieur). Par conséquent, nous pouvons utiliser le test de Wilcoxon pour analyser nos données.
Notez que, dans le cas où vos données ne sont pas distribuées symétriquement, vous pouvez envisager d’effectuer le test des signes, au lieu d’exécuter le test des rangs signés de Wilcoxon.
Le test des signes ne suppose pas l’hypothèse d’une distribution symétrique. Cependant, il sera probablement moins puissant que le test de Wilcoxon.
Calculs
Nous voulons savoir si le poids médian des souris diffère de 25g (test bilatéral) ?
stat.test <- mice %>% wilcox_test(weight ~ 1, mu = 25)
stat.test## # A tibble: 1 x 6
## .y. group1 group2 n statistic p
## * <chr> <chr> <chr> <int> <dbl> <dbl>
## 1 weight 1 null model 10 0 0.00195Notez que, pour calculer le test de Wilcoxon unilatéral, vous pouvez spécifier l’option alternative, dont les valeurs possibles peuvent être “greater”, “less” ou “two.sided”.
Taille de l’effet
Nous allons utiliser la fonction R wilcox_effsize() [rstatix]. Il nécessite le paquet coin pour calculer la statistique Z.
mice %>% wilcox_effsize(weight ~ 1, mu = 25)## # A tibble: 1 x 6
## .y. group1 group2 effsize n magnitude
## * <chr> <chr> <chr> <dbl> <int> <ord>
## 1 weight 1 null model 0.886 10 largeUne grande taille d’effet est détectée, r = 0,89.
Rapporter
Nous pourrions rapporter le résultat comme suit:
Un test de Wilcoxon a été effectué pour déterminer si le poids médian des souris recrutées était différent du poids médian normal de la population (25 g).
La valeur du poids des souris était distribuée de façon à peu près symétrique, comme l’indique l’histogramme avec la courbe de densité superposée.
Le poids médian mesuré chez la souris (19,8) était statistiquement significativement inférieur au poids médian de la population de 25 g (p = 0,002, taille de l’effet r = 0,89).
Créer un box plot avec p-value:
bxp +
labs(subtitle = get_test_label(stat.test, detailed = TRUE))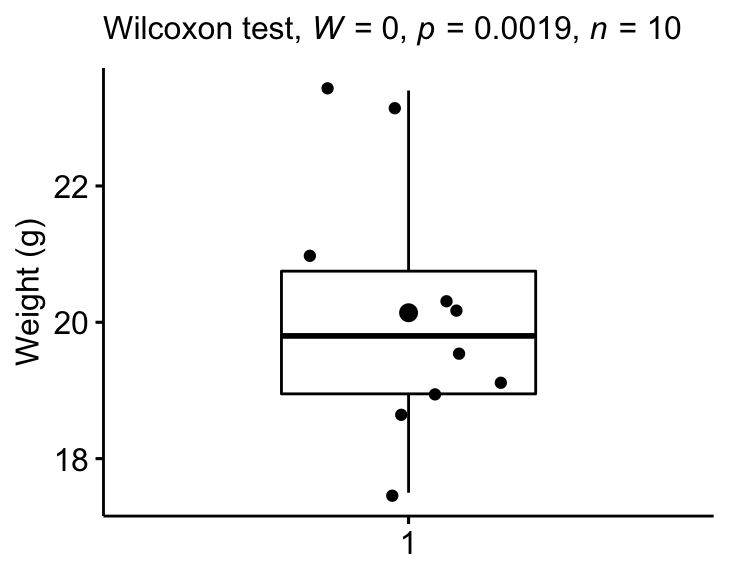
Créer un graphe de densité avec p-value:
- La ligne rouge correspond à la médiane observée
- La ligne bleue correspond à la médiane théorique
ggdensity(mice, x = "weight", rug = TRUE, fill = "lightgray") +
scale_x_continuous(limits = c(15, 27)) +
stat_central_tendency(type = "median", color = "red", linetype = "dashed") +
geom_vline(xintercept = 25, color = "blue", linetype = "dashed") +
labs(subtitle = get_test_label(stat.test, detailed = TRUE))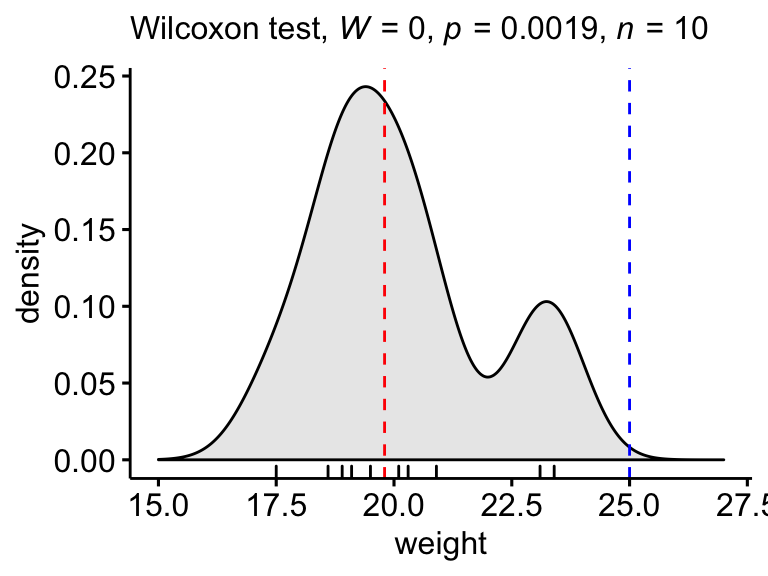
Test de la somme des rangs de Wilcoxon
Le test de la somme des rangs de Wilcoxon est une alternative non paramétrique au test t-test indépendant pour comparer deux groupes d’échantillons indépendants, dans le cas où les données ne sont pas normalement distribuées.
Synonyme : Test de Man-Whitney, test de Man-Whitney U, test de Wilcoxon-Mann-Whitney et test de Wilcoxon à deux échantillons.
Données de démonstration
Jeu de données de démonstration : genderweight [package datarium] contenant le poids de 40 individus (20 femmes et 20 hommes).
Charger les données et afficher quelques lignes aléatoires par groupes:
# Charger les données
data("genderweight", package = "datarium")
# Afficher un échantillon des données par groupe
set.seed(123)
genderweight %>% sample_n_by(group, size = 2)## # A tibble: 4 x 3
## id group weight
## <fct> <fct> <dbl>
## 1 6 F 65.0
## 2 15 F 65.9
## 3 29 M 88.9
## 4 37 M 77.0Statistiques descriptives
Calculer quelques statistiques sommaires par groupe : médiane et écart interquartile.
genderweight %>%
group_by(group) %>%
get_summary_stats(weight, type = "median_iqr")## # A tibble: 2 x 5
## group variable n median iqr
## <fct> <chr> <dbl> <dbl> <dbl>
## 1 F weight 20 62.9 2.33
## 2 M weight 20 86.3 4.59Visualisation
Visualiser les données à l’aide de box plots. Graphique du poids par groupes.
bxp <- ggboxplot(
genderweight, x = "group", y = "weight",
ylab = "Weight", xlab = "Groups", add = "jitter"
)
bxp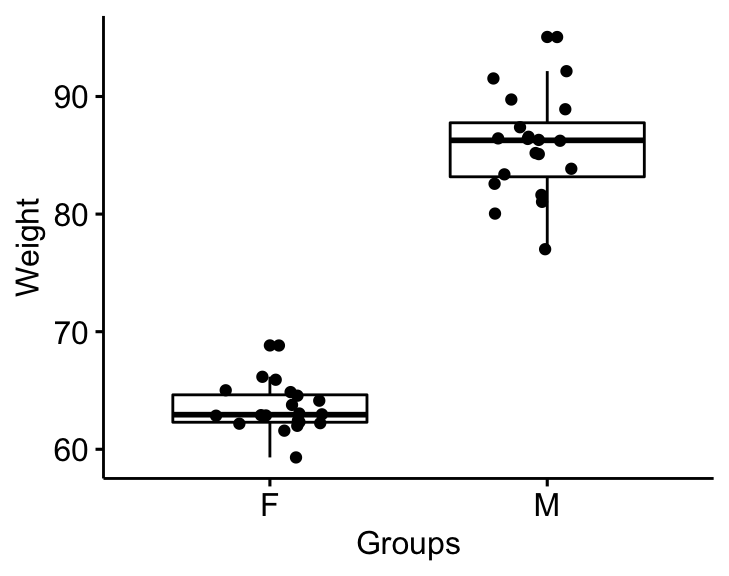
Calculs
Question : Y a-t-il une différence significative entre le poids médian des femmes et celui des hommes ?
stat.test <- genderweight %>%
wilcox_test(weight ~ group) %>%
add_significance()
stat.test## # A tibble: 1 x 8
## .y. group1 group2 n1 n2 statistic p p.signif
## <chr> <chr> <chr> <int> <int> <dbl> <dbl> <chr>
## 1 weight F M 20 20 0 1.45e-11 ****Taille de l’effet
genderweight %>% wilcox_effsize(weight ~ group)## # A tibble: 1 x 7
## .y. group1 group2 effsize n1 n2 magnitude
## * <chr> <chr> <chr> <dbl> <int> <int> <ord>
## 1 weight F M 0.855 20 20 largeUne grande taille d’effet est détectée, r = 0,86.
Rapporter
Nous pourrions rapporter le résultat comme suit:
Le poids médian dans le groupe des femmes était de 62,9 (IQR = 2,33), tandis que celui des hommes était de 86,3 (IQR = 4,59). Le test de Wilcoxon a montré que la différence était significative (p < 0,0001, taille de l’effet r = 0,86).
stat.test <- stat.test %>% add_xy_position(x = "group")
bxp +
stat_pvalue_manual(stat.test, tip.length = 0) +
labs(subtitle = get_test_label(stat.test, detailed = TRUE))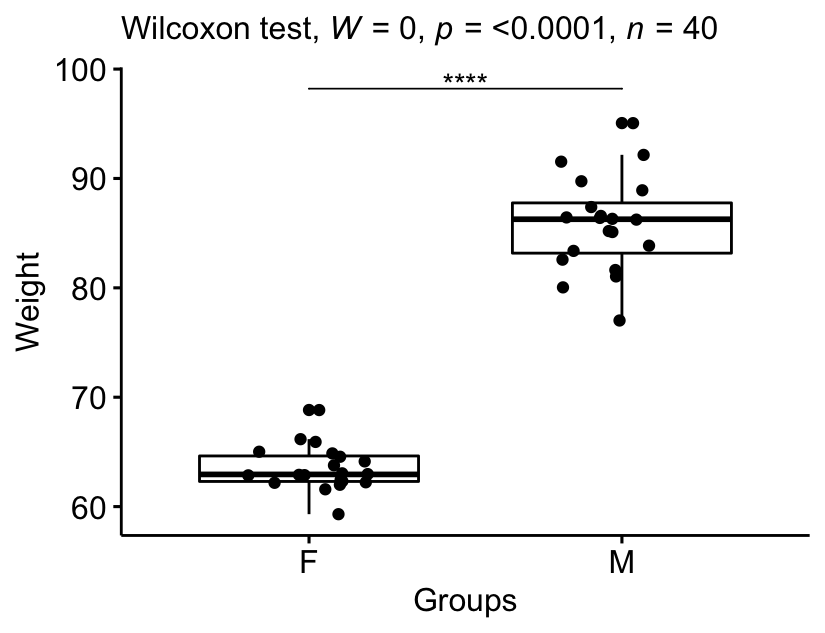
Test des rangs signés de Wilcoxon sur échantillons appariés
Le test des rangs signés de Wilcoxon sur échantillons appariés est une alternative non paramétrique au test t sur échantillons appariés pour comparer les données appariés. Il est utilisé lorsque les données ne sont pas distribuées normalement.
Données de démonstration
Ici, nous utiliserons un jeu de données de démonstration mice2 [package datarium], qui contient le poids de 10 souris avant et après le traitement.
# Format large
data("mice2", package = "datarium")
head(mice2, 3)## id before after
## 1 1 187 430
## 2 2 194 404
## 3 3 232 406# Transformez en données longues :
# rassembler les valeurs de `before` (avant) et `after` (après) dans la même colonne
mice2.long <- mice2 %>%
gather(key = "group", value = "weight", before, after)
head(mice2.long, 3)## id group weight
## 1 1 before 187
## 2 2 before 194
## 3 3 before 232Statistiques descriptives
Calcul de certaines statistiques sommaires par groupes : médiane et intervalle interquartile (IQR).
mice2.long %>%
group_by(group) %>%
get_summary_stats(weight, type = "median_iqr")## # A tibble: 2 x 5
## group variable n median iqr
## <chr> <chr> <dbl> <dbl> <dbl>
## 1 after weight 10 405 28.3
## 2 before weight 10 197. 19.2Visualisation
bxp <- ggpaired(mice2.long, x = "group", y = "weight",
order = c("before", "after"),
ylab = "Weight", xlab = "Groups")
bxp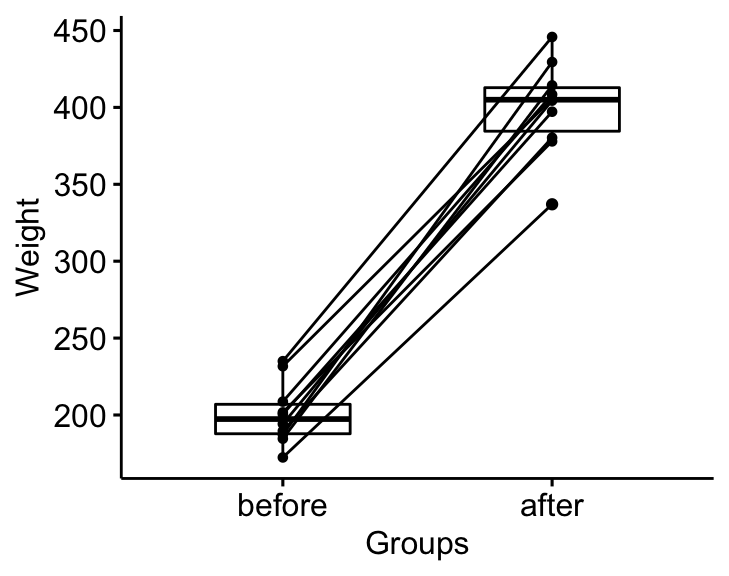
Hypothèses et tests préliminaires
Le test suppose que les différences entre les échantillons appariés doivent être distribuées symétriquement autour de la médiane.
Calculer les différences entre les paires et créer des histogrammes:
mice2 <- mice2 %>% mutate(differences = after - before)
gghistogram(mice2, x = "differences", y = "..density..",
fill = "steelblue",bins = 5, add_density = TRUE)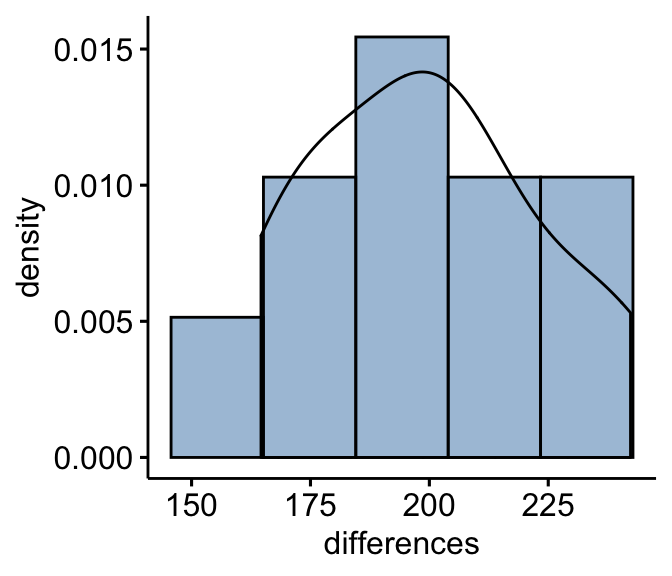
D’après le graphique ci-dessus, on peut voir que les données sur les différences sont à peu près symétriques (il ne faut pas s’attendre à ce qu’elles soient parfaites, surtout lorsque le nombre d’échantillons dans votre étude est plus petit). Par conséquent, nous pouvons utiliser le test de Wilcoxon pour analyser nos données.
Notez que, dans le cas où vos données ne sont pas distribuées symétriquement, vous pouvez envisager d’effectuer le test des signes, au lieu d’exécuter le test des rangs signés de Wilcoxon.
Le test des signes ne suppose pas l’hypothèse d’une distribution symétrique. Cependant, il sera probablement moins puissant que le test de Wilcoxon.
Calculs
Question : Y a-t-il des changements significatifs dans le poids des souris après le traitement ?
stat.test <- mice2.long %>%
wilcox_test(weight ~ group, paired = TRUE) %>%
add_significance()
stat.test## # A tibble: 1 x 8
## .y. group1 group2 n1 n2 statistic p p.signif
## <chr> <chr> <chr> <int> <int> <dbl> <dbl> <chr>
## 1 weight after before 10 10 55 0.00195 **Taille de l’effet
mice2.long %>%
wilcox_effsize(weight ~ group, paired = TRUE)## # A tibble: 1 x 7
## .y. group1 group2 effsize n1 n2 magnitude
## * <chr> <chr> <chr> <dbl> <int> <int> <ord>
## 1 weight after before 0.886 10 10 largeUne grande taille d’effet est détectée, r = 0,89.
Rapporter
D’après les résultats ci-dessus, on peut conclure que le poids médian des souris avant traitement est significativement différent du poids médian après traitement avec une p-value = 0,002, taille de l’effet r = 0,89.
stat.test <- stat.test %>% add_xy_position(x = "group")
bxp +
stat_pvalue_manual(stat.test, tip.length = 0) +
labs(subtitle = get_test_label(stat.test, detailed= TRUE))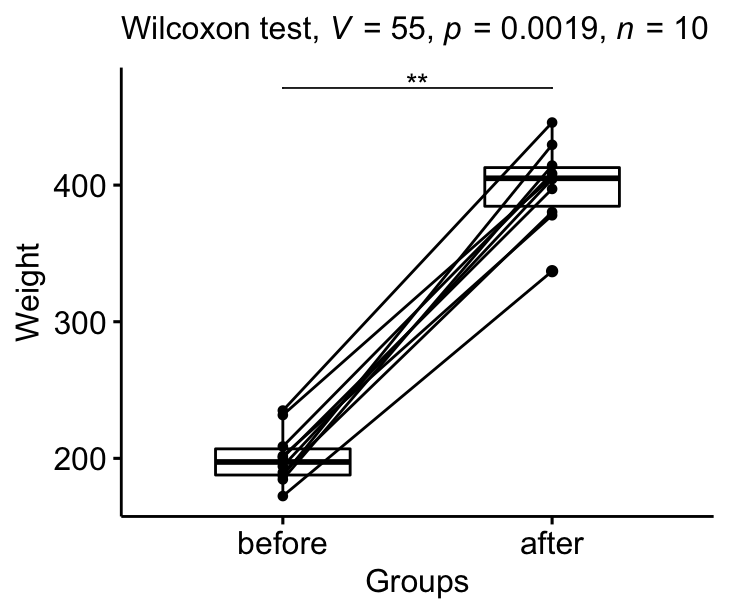
Résumé
Ce chapitre décrit comment comparer deux moyennes dans R en utilisant le test de Wilcoxon, qui est une alternative non paramétrique du test t.
Les codes R à démarrage rapide, pour calculer les différents tests de Wilcoxon, sont les suivants:
# Test des rangs signés de Wilcoxon sur échantillon unique
mice %>% wilcox_test(weight ~ 1, mu = 25)
# Test de la somme des rangs de Wilcoxon : échantillons indépendants
genderweight %>% wilcox_test(weight ~ group)
# Test des rangs signés de Wilcoxon sur échantillons appariés
mice2.long %>% wilcox_test(weight ~ group, paired = TRUE)Notez que, pour calculer les tests de Wilcoxon unilatéraux, vous pouvez spécifier l’option alternative, dont les valeurs possibles peuvent être “greater”, “less” ou “two.sided”.
Version:
 English
English



No Comments