Le test-t apparié est utilisé pour comparer les moyennes de deux groupes d’échantillons apparentés. En d’autres termes, il est utilisé dans une situation où vous avez deux valeurs (c’est-à-dire une paire de valeurs) pour les mêmes échantillons.
Il est également appelé:
- test-t dépendent,
- test-t pour échantillons dépendants,
- test-t pour mesures répétées,
- test-t pour échantillons reliés,
- test-t à deux échantillons appariés,
- test t pour échantillon apparié et
- test t pour moyennes dépendantes.
Par exemple, vous pouvez comparer le poids moyen de 20 souris avant et après le traitement. Les données contiennent 20 ensembles de valeurs avant traitement et 20 ensembles de valeurs après traitement provenant de la double mesure du poids d’une même souris. Dans de telles situations, le test t-test par paires peut être utilisé pour comparer les poids moyens avant et après le traitement.
La procédure de l’analyse du test t apparié est la suivante:
- Calculer la différence (\(d\)) entre chaque paire de valeur
- Calculer la moyenne (\(m\)) et l’écart-type (\(s\)) de \(d\)
- Comparer la différence moyenne à 0. S’il y a une différence significative entre les deux paires d’échantillons, alors la moyenne de d (\(m\)) devrait être loin de 0.
Le test t apparié ne peut être utilisé que lorsque la différence \(d\) est normalement distribuée. Cela peut être vérifié à l’aide du test de Shapiro-Wilk.
Dans ce chapitre, vous apprendrez la formule du test t apparié, ainsi que, comment:
- Calculer le test t apparié dans R. La fonction
t_test()[paquet rstatix], qui est compatible avec les pipes, sera utilisée. - Vérifier les hypothèses du test t apparié
- Calculez et rapportez la taille de l’effet du test t apparié en utilisant le d de Cohen. La statistique “d” redéfinit la différence de moyennes comme le nombre d’écarts-types qui sépare ces moyennes. Les tailles d’effet conventionnelles des tests T, proposées par Cohen, sont : 0,2 (petit effet), 0,5 (effet modéré) et 0,8 (effet important) (Cohen 1998).
Sommaire:
Livre Apparenté
Pratique des Statistiques dans R II - Comparaison de Groupes: Variables NumériquesPrérequis
Assurez-vous d’avoir installé les paquets R suivants:
tidyversepour la manipulation et la visualisation des donnéesggpubrpour créer facilement des graphiques prêts à la publicationrstatixcontient des fonctions R facilitant les analyses statistiques.datarium: contient les jeux de données requis pour ce chapitre.
Commencez par charger les packages requis suivants:
library(tidyverse)
library(ggpubr)
library(rstatix)Questions de recherche
Les questions de recherche typiques sont:
- si la différence moyenne (\(m\)) est égale à 0 ?
- si la différence moyenne (\(m\)) est inférieure à 0 ?
- si la différence moyenne (\(m\)) est plus grande que 0 ?
hypothèses statistiques
En statistique, on peut définir l’hypothèse nulle correspondante (\(H_0\)) comme suit:
- \(H_0 : m = 0\)
- H_0 : m 0$
- H_0 : m 0$
Les hypothèses alternatives correspondantes (\(H_a\)) sont les suivantes:
- \(H_a : m \ne 0\) (différent)
- \(H_a : m > 0\) (plus grand ou “greater” en anglais)
- \(H_a : m < 0\) (plus petit ou “lesser” en anglais)
Notez que:
- Les hypothèses 1) sont appelées tests bilatéraux
- Les hypothèses 2) et 3) sont appelées tests unilatéraux
Formule
La valeur statistique du test t apparié peut être calculée à l’aide de la formule suivante:
\[
t = \frac{m}{s/\sqrt{n}}
\]
où,
mest la moyenne des différencesnest la taille de l’échantillon (c.-à-d. la taille de d).sest l’écart-type de d
On peut calculer la p-value correspondant à la valeur absolue de la statistique du t-test (|t|) pour les degrés de liberté (df) : \(df = n - 1\).
Si la p-value est inférieure ou égale à 0,05, on peut conclure que la différence entre les deux échantillons appariés est significativement différente.
Données de démonstration
Ici, nous utiliserons un jeu de données de démonstration mice2 [package datarium], qui contient le poids de 10 souris avant et après le traitement.
# Format large
data("mice2", package = "datarium")
head(mice2, 3)## id before after
## 1 1 187 430
## 2 2 194 404
## 3 3 232 406# Transformez en données longues :
# rassembler les valeurs de `before` (avant) et `after` (après) dans la même colonne
mice2.long <- mice2 %>%
gather(key = "group", value = "weight", before, after)
head(mice2.long, 3)## id group weight
## 1 1 before 187
## 2 2 before 194
## 3 3 before 232Statistiques descriptives
Calculer quelques statistiques descriptives (moyenne et sd) par groupe:
mice2.long %>%
group_by(group) %>%
get_summary_stats(weight, type = "mean_sd")## # A tibble: 2 x 5
## group variable n mean sd
## <chr> <chr> <dbl> <dbl> <dbl>
## 1 after weight 10 400. 30.1
## 2 before weight 10 201. 20.0Visualisation
bxp <- ggpaired(mice2.long, x = "group", y = "weight",
order = c("before", "after"),
ylab = "Weight", xlab = "Groups")
bxp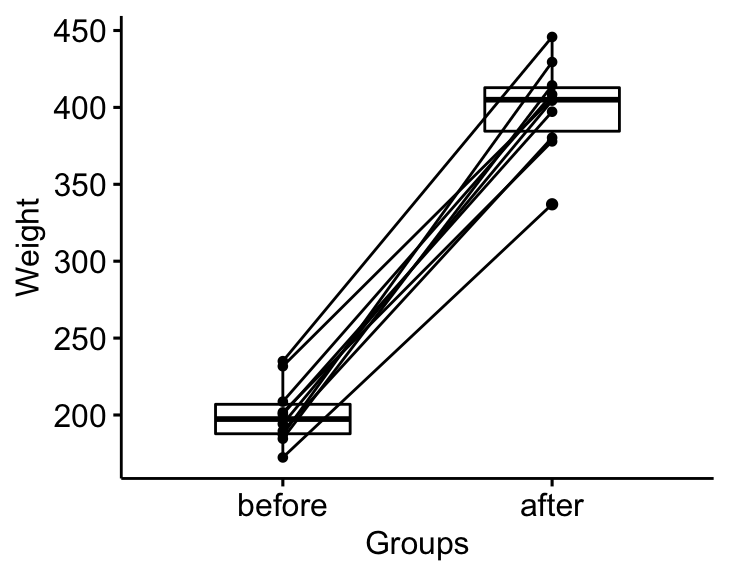
Hypothèses et tests préliminaires
Le test t des échantillons appariés suppose les caractéristiques suivantes au sujet des données:
- les deux groupes sont appariés. Dans notre exemple, c’est le cas puisque les données ont été recueillies en mesurant deux fois le poids des mêmes souris.
- Aucune valeur aberrante significative dans la différence entre les deux groupes appariés
- Normalité. la différence des paires suit une distribution normale.
Dans cette section, nous effectuerons quelques tests préliminaires pour vérifier si ces hypothèses sont respectées.
Tout d’abord, commencez par calculer la différence entre les groupes:
mice2 <- mice2 %>% mutate(differences = before - after)
head(mice2, 3)## id before after differences
## 1 1 187 430 -242
## 2 2 194 404 -210
## 3 3 232 406 -174Identifier les valeurs aberrantes
Les valeurs aberrantes peuvent être facilement identifiées à l’aide des méthodes boxplot, implémentées dans la fonction R identify_outliers() [paquet rstatix].
mice2 %>% identify_outliers(differences)## [1] id before after differences is.outlier is.extreme
## <0 rows> (or 0-length row.names)Il n’y avait pas de valeurs extrêmes aberrantes.
Notez que, dans le cas où vous avez des valeurs extrêmes aberrantes, cela peut être dû à : 1) erreurs de saisie de données, erreurs de mesure ou valeurs inhabituelles.
Vous pouvez quand même inclure la valeur aberrante dans l’analyse si vous ne croyez pas que le résultat sera affecté de façon substantielle. Cela peut être évalué en comparant le résultat du test t avec et sans la valeur aberrante.
Il est également possible de conserver les valeurs aberrantes dans les données et d’effectuer un test Wilcoxon ou un test t robuste en utilisant le progiciel WRS2.
Vérifier l’hypothèse de normalité
L’hypothèse de normalité peut être vérifiée en calculant le test de Shapiro-Wilk pour chaque groupe. Si les données sont normalement distribuées, la p-value doit être supérieure à 0,05.
mice2 %>% shapiro_test(differences) ## # A tibble: 1 x 3
## variable statistic p
## <chr> <dbl> <dbl>
## 1 differences 0.968 0.867D’après le résultat, les deux p-values sont supérieures au seuil de significativité 0,05, ce qui indique que la distribution des données n’est pas significativement différente de la distribution normale. En d’autres termes, nous pouvons supposer que la normalité.
Vous pouvez également créer des QQ plots pour chaque groupe. Le graphique QQ plot dessine la corrélation entre une donnée définie et la distribution normale.
ggqqplot(mice2, "differences")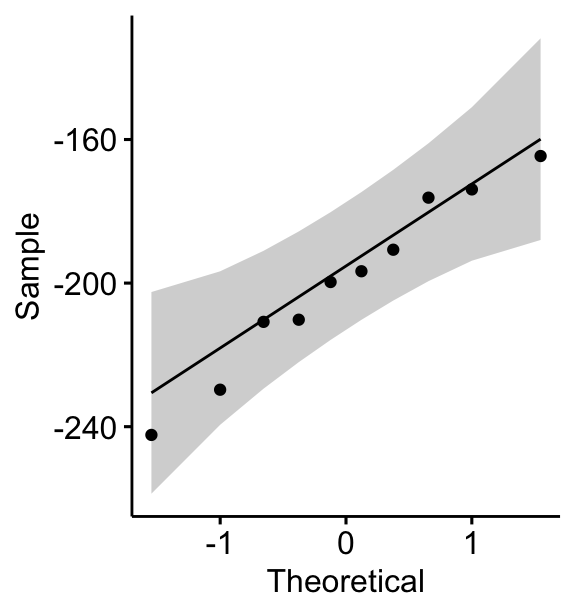
Tous les points se situent approximativement le long de la ligne de référence (45 degrés), pour chaque groupe. Nous pouvons donc supposer la normalité des données.
Notez que, si la taille de votre échantillon est supérieure à 50, le graphique de normalité QQ plot est préféré parce qu’avec des échantillons de plus grande taille, le test de Shapiro-Wilk devient très sensible même à un écart mineur par rapport à la distribution normale.
Dans le cas où les données ne sont pas normalement distribuées, il est recommandé d’utiliser le test de Wilcoxon non paramétrique.
Calculs
Nous voulons savoir s’il y a une différence significative dans les poids moyens après le traitement ?
Nous allons utiliser la fonction t_test() [package rstatix], facile d’utilisation, un emballage autour de la fonction de base R t.test().
stat.test <- mice2.long %>%
t_test(weight ~ group, paired = TRUE) %>%
add_significance()
stat.test## # A tibble: 1 x 9
## .y. group1 group2 n1 n2 statistic df p p.signif
## <chr> <chr> <chr> <int> <int> <dbl> <dbl> <dbl> <chr>
## 1 weight after before 10 10 25.5 9 0.00000000104 ****Les résultats ci-dessus montrent les composantes suivantes:
.y.: la variable y utilisée dans le test.group1,group2: les groupes comparés dans les tests par paires.statistic: Statistique de test utilisée pour calculer la p-value.df: degrés de liberté.p: p-value.
Notez que, vous pouvez obtenir un résultat détaillé en spécifiant l’option detailed = TRUE.
Pour calculer un t-test pairé unilatéral, vous pouvez spécifier l’option alternative comme suit.
- si vous voulez tester si le poids moyen avant traitement est inférieur au poids moyen après traitement, saisissez ceci:
mice2.long %>%
t_test(weight ~ group, paired = TRUE, alternative = "less")- Ou, si vous voulez tester si le poids moyen avant traitement est supérieur au poids moyen après traitement, saisissez ceci
mice2.long %>%
t_test(weight ~ group, paired = TRUE, alternative = "greater")Taille de l’effet
La taille de l’effet d’un test t pour échantillons appariés peut être calculée en divisant la différence moyenne par l’écart-type de la différence, comme indiqué ci-dessous.
La formule du d de Cohen:
\[
d = \frac{mean_D}{SD_D}
\]
Où D est la différence entre les valeurs des échantillons appariés.
Calculs:
mice2.long %>% cohens_d(weight ~ group, paired = TRUE)## # A tibble: 1 x 7
## .y. group1 group2 effsize n1 n2 magnitude
## * <chr> <chr> <chr> <dbl> <int> <int> <ord>
## 1 weight after before 8.08 10 10 largeLa taille de l’effet est importante, d de Cohen = 8,07.
Rapporter
Nous pourrions rapporter les résultats comme suit : Le poids moyen des souris a augmenté de façon significative après le traitement, t(9) = 25,5, p < 0,0001, d = 8,07.
stat.test <- stat.test %>% add_xy_position(x = "group")
bxp +
stat_pvalue_manual(stat.test, tip.length = 0) +
labs(subtitle = get_test_label(stat.test, detailed= TRUE))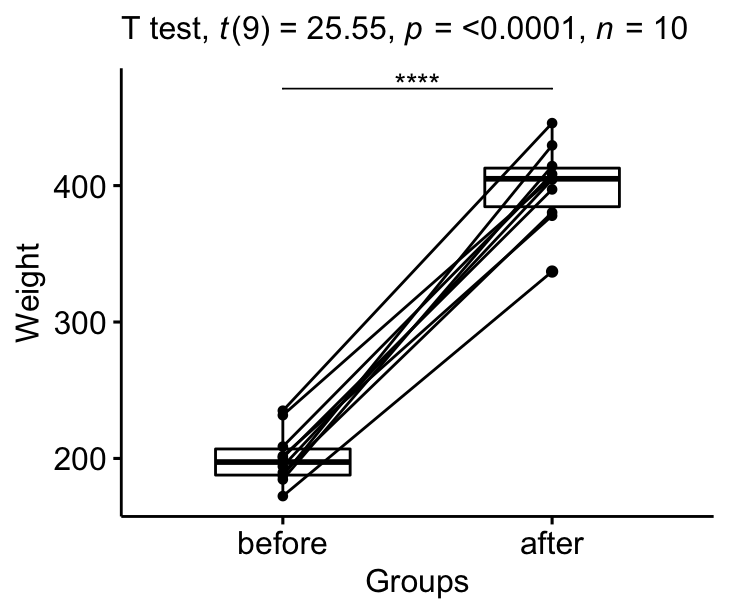
Résumé
Cet article décrit la formule et les principes de base du test t apparié ou du test t dépendant. Des exemples de codes R sont fournis pour vérifier les hypothèses, calculer le test et la taille de l’effet, interpréter et communiquer les résultats.
References
Cohen, J. 1998. Statistical Power Analysis for the Behavioral Sciences. 2nd ed. Hillsdale, NJ: Lawrence Erlbaum Associates.
Version:
 English
English



No Comments